Esta es la aplicación de Linux llamada raxmlGUI cuya última versión se puede descargar como raxmlGUI1.5b3.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada raxmlGUI con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie el emulador en línea OnWorks Linux o Windows en línea o el emulador en línea MACOS desde este sitio web.
- 5. Desde el SO OnWorks Linux que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación, instálala y ejecútala.
SCREENSHOTS
Ad
raxmlGUI
DESCRIPCIÓN
NOTA DE PUBLICACIÓN: Obtenga raxmlGUI 2.0 en la NUEVA UBICACIÓN DEL PROYECTO: https://antonellilab.github.io/raxmlGUI/raxmlGUI es una interfaz gráfica de usuario para RAxML, uno de los software más populares y ampliamente utilizados para la inferencia filogenética con máxima verosimilitud.
Una interfaz gráfica fácil de usar para análisis filogenéticos utilizando RAxML (Stamatakis, 2006). Por favor cite: Silvestro, Michalak (2012) - raxmlGUI: una interfaz gráfica para RAxML. Diversidad y evolución de organismos 12, 335-337. DOI: 10.1007 / s13127-011-0056-0
Caracteristicas
- * NUEVA VERSIÓN 2.0 AHORA DISPONIBLE: https://antonellilab.github.io/raxmlGUI/*
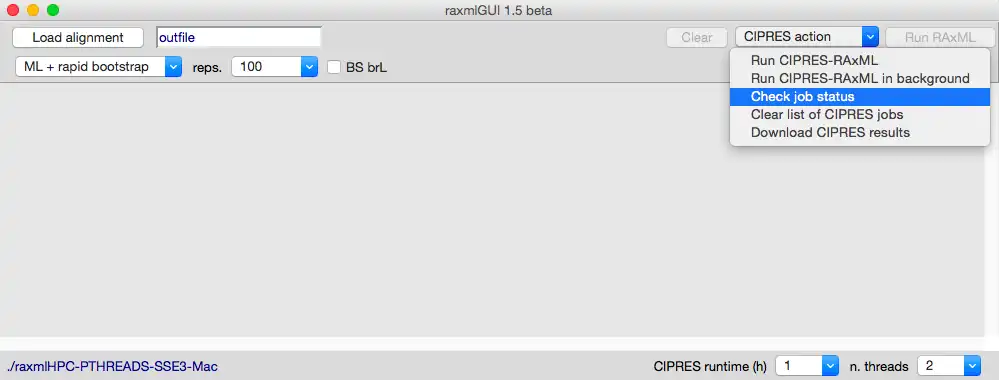
- * la versión 1.5 * agrega la posibilidad de ejecutar análisis a través de CIPRES Science Gateway
- La GUI incluye ejecutables RAxML
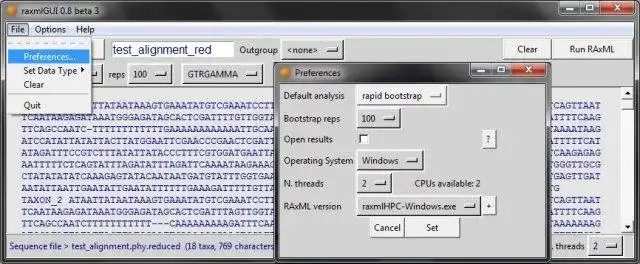
- Compatibilidad multiplataforma (Mac OS, Windows, Linux)
- Requiere Python 2.6, 2.7
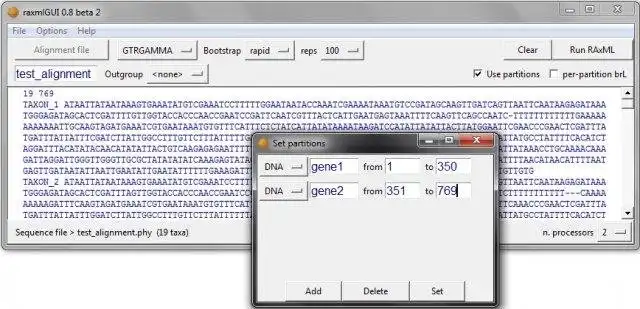
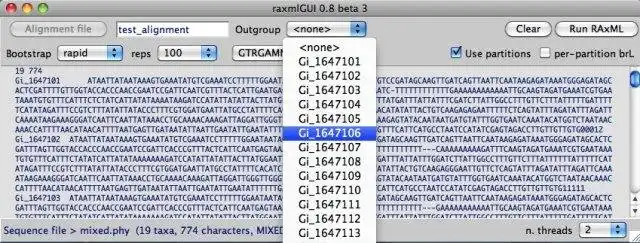
- Interfaz intuitiva para la partición de datos y la selección de grupos externos
- Selección de modelos para alineamientos de ADN, aminoácidos, binarios y multiestado
- Conversión automática de NEXUS a PHYLIP (y viceversa)
- Importar archivos FASTA
- Reconstrucción del estado ancestral
- Función "Bootstopping" (Pattengale et al. 2010)
- Exclusión interactiva de sitios
- Combinando múltiples alineaciones con partición automática
- Definición interactiva de restricciones topológicas
- Distancias por pares de Robinson-Foulds
- Probabilidades por sitio
Audiencia
Ciencia / Investigación
Interfaz de usuario
Tk
Lenguaje de programación
Python
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/raxmlgui/. Se ha alojado en OnWorks para poder ejecutarlo online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.