Esta es la aplicación de Linux llamada gsasnp2 cuya última versión se puede descargar como gsasnp2-linux-cmd-ubuntu.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada gsasnp2 con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie el emulador en línea OnWorks Linux o Windows en línea o el emulador en línea MACOS desde este sitio web.
- 5. Desde el SO OnWorks Linux que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación, instálala y ejecútala.
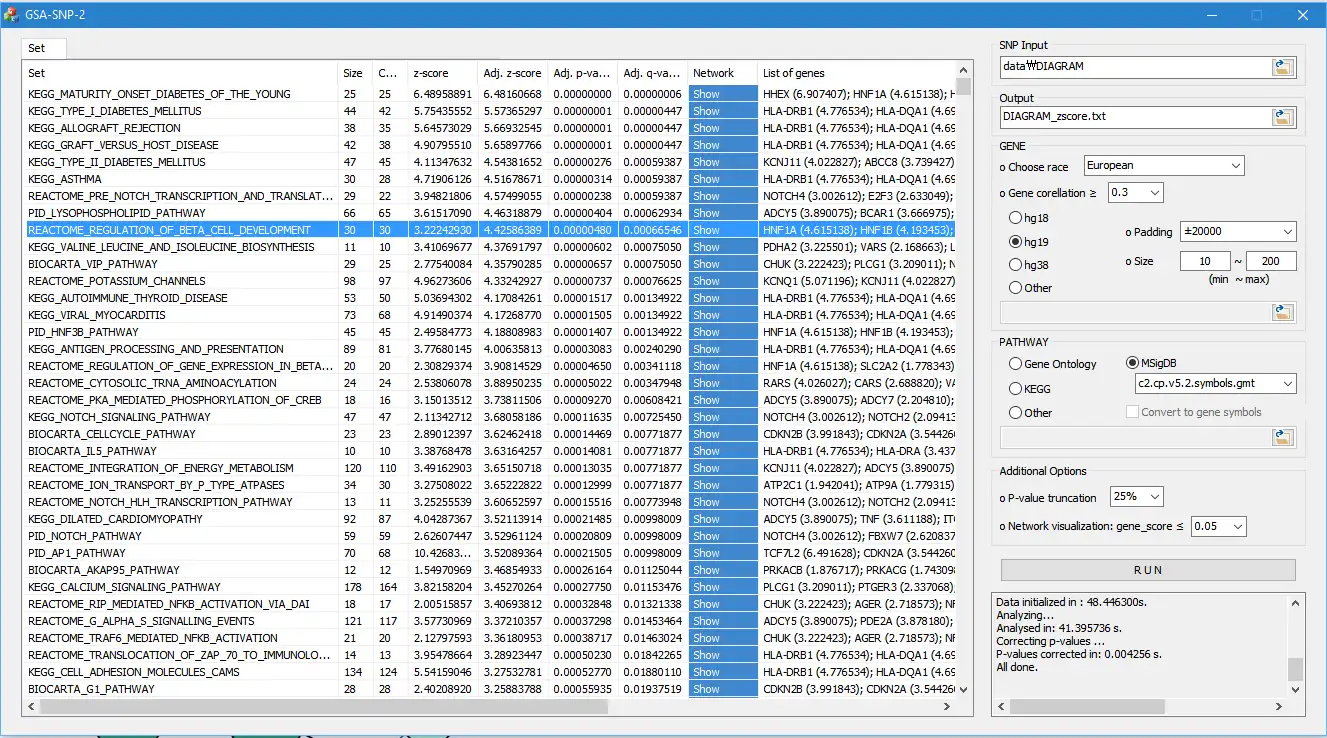
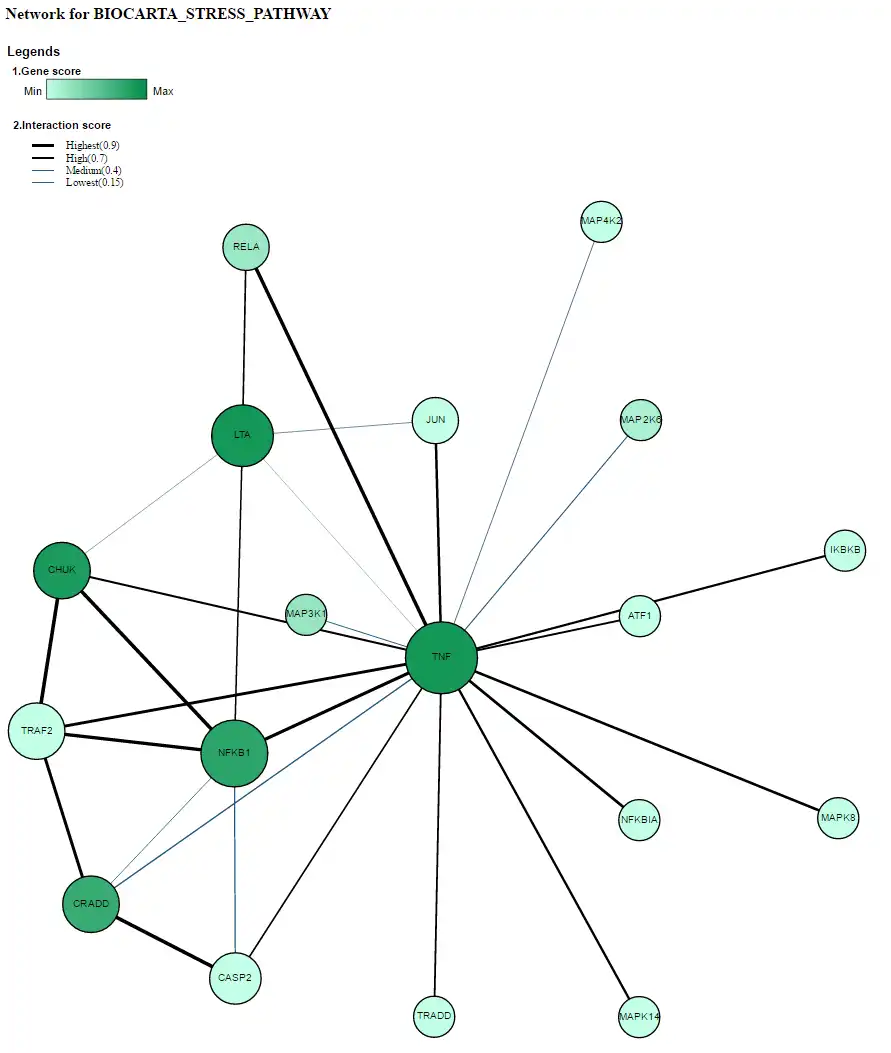
SCREENSHOTS
Ad
gsasnp2
DESCRIPCIÓN
* GSA-SNP2 es un sucesor de GSA-SNP (Nam et al. 2010, problema del servidor web NAR). GSA-SNP2 acepta datos de resumen de GWAS humanos (números rs, valores p) o valores p genéticos y genera conjuntos de genes de la vía 'enriquecidos' con genes asociados con el fenotipo dado. También proporciona redes de interacción de proteínas tanto locales como globales en las vías asociadas.
* Artículo: SYoon, HCTNguyen, YJYoo, JKim, BBaik, SKim, JKim, SKim, DNam, "Enriquecimiento eficiente de vías y análisis de redes de datos resumidos de GWAS usando GSA-SNP2", Investigación de ácidos nucleicos, vol. 46 (10), e60 (2018).
* ID de PubMed: 29562348
* DOI: 10.1093 / nar / gky175
-> POR FAVOR, MUEVA O HAGA UNA COPIA DE LA CARPETA DE 'DATOS' EN SU CARPETA DE PRUEBA INTENSIVA (POR EJEMPLO, CARPETA ESPECIFICADA DE LINUX, MAC O WINDOWS) PARA PERMITIR QUE EL PROGRAMA ENCUENTRE LOS DATOS PREDEFINIDOS.
* ACTUALIZAR NOTA:
-> 1 de septiembre de 2020: agregue una actualización para Ubuntu-20.04. Necesitará la biblioteca Boost instalada (sudo apt-get install libboost-all-dev)
-> 7 de marzo de 2018: revisar los términos del encabezado en el archivo de salida
Caracteristicas
- 1 / 'CONTROL DE ERRORES TIPO I DECENTE' logrado mediante los dos procesos siguientes: A) Las puntuaciones de los genes se 'ajustan' al número de SNP asignados a cada gen utilizando una curva de tendencia spline cúbica monótona. B) Se eliminaron los genes adyacentes con altas correlaciones entre genes dentro de cada vía.
- 2 / 'ALTA POTENCIA Y COMPUTACIÓN RÁPIDA' basado en el modelo de conjunto aleatorio
- 3 / 'SIN PARÁMETRO LIBRE CRÍTICO'
- Se visualizaron 4 / 'REDES DE INTERACCIÓN DE PROTEÍNAS' entre los genes miembros para las vías significativas. Esta función permite al usuario priorizar las subredes centrales dentro y a través de las rutas importantes. Actualmente se proporcionan las redes STRING e HIPPIE
- 5 / 'FÁCIL DE USAR': Solo requiere datos resumidos de GWAS (o valores p de genes) y solo toma uno o dos minutos obtener resultados. Otras potentes herramientas de ruta autónomas también requieren la entrada de correlación SNP y requieren mucho más tiempo. El usuario también puede cargar sus propios conjuntos de genes de vías y redes de interacción de proteínas.
Interfaz de usuario
Win32 (MS Windows), línea de comandos
Lenguaje de programación
C ++, PHP, JavaScript
Categorías
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/gsasnp2/. Se ha alojado en OnWorks para poder ejecutarlo online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.