Esta es la aplicación de Linux llamada PARSEC - PAtteRn SEarch / Context para ejecutar en Linux en línea, cuya última versión se puede descargar como PARSEC_Deployment_Package_v2.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada PARSEC - PAtteRn SEarch / Context para ejecutar en Linux en línea con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie el emulador en línea OnWorks Linux o Windows en línea o el emulador en línea MACOS desde este sitio web.
- 5. Desde el SO OnWorks Linux que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación, instálala y ejecútala.
SCREENSHOTS
Ad
PARSEC - BÚSQUEDA DE PATRÓN / Contexto para ejecutar en Linux en línea
DESCRIPCIÓN
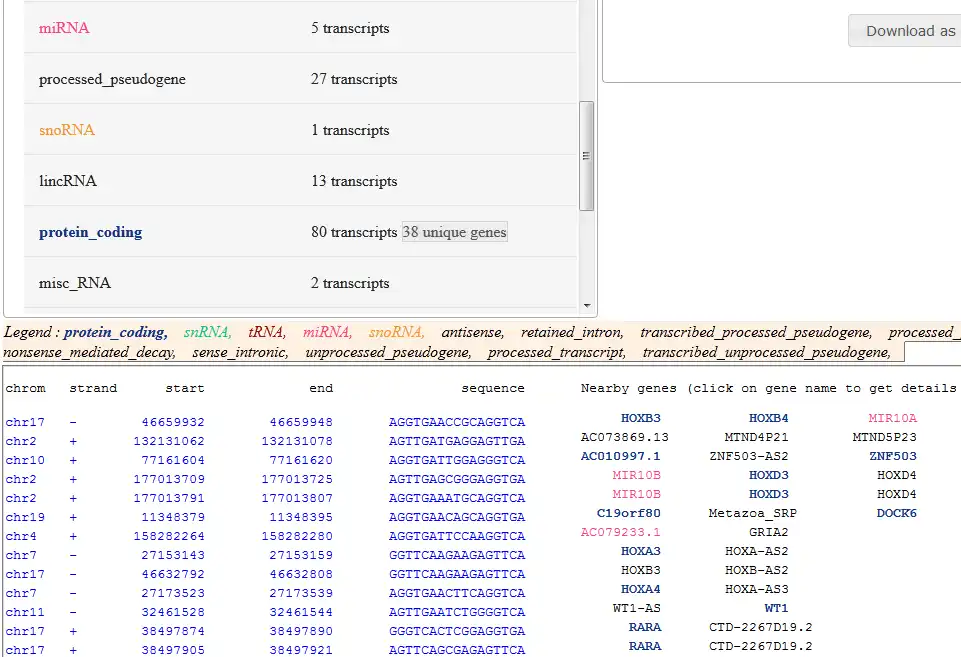
La caracterización de sitios genómicos es un desafío importante en la comprensión y explotación de datos de secuenciación de próxima generación. La mayoría de los sitios genómicos están representados por motivos cortos y degenerados con una distribución dispersa y, a veces, con función biológica (por ejemplo, regulación de la expresión génica, patrones de empalme o señales epigenéticas). Estos motivos están asociados con una gran cantidad de ruido y, por lo tanto, el desarrollo de una plataforma computacional para la detección precisa de sitios genómicos requiere la integración de varios datos biológicos a gran escala para filtrar los falsos positivos.PARSEC representa una solución intuitiva, modular (fácilmente ampliable) y todo en uno para la integración eficiente de gran cantidad de información genómica diversa con el fin de realizar la localización y caracterización no lineal de sitios biológicos en un entorno fácil de usar.
Consulte la wiki para conocer los requisitos de hardware y los navegadores compatibles.
Audiencia
Ciencia / Investigación
Interfaz de usuario
Basado en la Web
Lenguaje de programación
C ++, Java
Entorno de base de datos
JDBC, MySQL, PostgreSQL (pgsql)
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/genomicparsec/. Se ha alojado en OnWorks para poder ejecutarlo online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.