Esta es la aplicación de Windows llamada Calis-p para ejecutarse en Windows en línea sobre Linux en línea, cuya última versión se puede descargar como calis-p-0.1.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada Calis-p para ejecutarla en Windows en línea sobre Linux en línea con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie cualquier emulador en línea de OS OnWorks desde este sitio web, pero mejor emulador en línea de Windows.
- 5. Desde el sistema operativo OnWorks Windows que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación e instálala.
- 7. Descargue Wine desde los repositorios de software de sus distribuciones de Linux. Una vez instalada, puede hacer doble clic en la aplicación para ejecutarla con Wine. También puedes probar PlayOnLinux, una elegante interfaz sobre Wine que te ayudará a instalar programas y juegos populares de Windows.
Wine es una forma de ejecutar software de Windows en Linux, pero no requiere Windows. Wine es una capa de compatibilidad de Windows de código abierto que puede ejecutar programas de Windows directamente en cualquier escritorio de Linux. Esencialmente, Wine está tratando de volver a implementar una cantidad suficiente de Windows desde cero para poder ejecutar todas esas aplicaciones de Windows sin necesidad de Windows.
SCREENSHOTS
Ad
Calis-p para ejecutarse en Windows en línea sobre Linux en línea
DESCRIPCIÓN
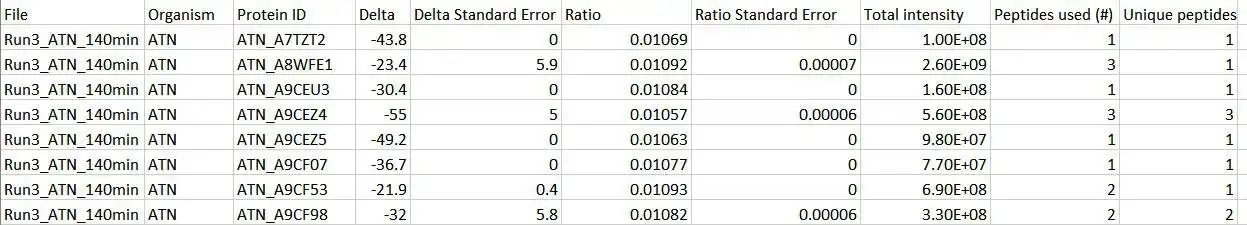
Calis-p (El enfoque CALgary de los isótopos en proteómica) es un paquete de software para extraer directamente huellas dactilares de isótopos de carbono estables (SIF) de especies individuales en una comunidad microbiana a partir de un conjunto de datos metaproteómicos. Toma las tablas de coincidencia de espectro de péptidos (PSM) puntuados para las muestras y el material de calibración, y los datos de MS sin procesar en formato mzML como entrada. En el primer paso, el software encuentra los picos isotópicos para cada PSM y suma sus intensidades en una ventana de tiempo de retención específica. Los patrones isotópicos identificados (pares de valor m / z + intensidades sumadas) junto con la fórmula de suma de los péptidos identificados se informan y utilizan como entrada para el segundo paso de Calis-p para calcular los valores delta13C para todos los péptidos que pasan un conjunto de filtros, así como valores promedio delta13C y errores estándar para cada especie. Los valores de especies delta13C deben corregirse para el fraccionamiento de isótopos del instrumento aplicando la compensación determinada utilizando el material de referencia.Caracteristicas
- Los datos SIF se extraen directamente de un conjunto de datos metaproteómicos estándar
- Obtenga de manera eficiente y confiable valores de SIF para un gran número de especies en una muestra de comunidad microbiana de una manera de alto rendimiento.
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/calis-p/. Se ha alojado en OnWorks para poder ejecutarlo online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.