Esta es la aplicación de Windows llamada gsasnp2 cuya última versión se puede descargar como gsasnp2-linux-cmd-ubuntu.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada gsasnp2 con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie cualquier emulador en línea de OS OnWorks desde este sitio web, pero mejor emulador en línea de Windows.
- 5. Desde el sistema operativo OnWorks Windows que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación e instálala.
- 7. Descargue Wine desde los repositorios de software de sus distribuciones de Linux. Una vez instalada, puede hacer doble clic en la aplicación para ejecutarla con Wine. También puedes probar PlayOnLinux, una elegante interfaz sobre Wine que te ayudará a instalar programas y juegos populares de Windows.
Wine es una forma de ejecutar software de Windows en Linux, pero no requiere Windows. Wine es una capa de compatibilidad de Windows de código abierto que puede ejecutar programas de Windows directamente en cualquier escritorio de Linux. Esencialmente, Wine está tratando de volver a implementar una cantidad suficiente de Windows desde cero para poder ejecutar todas esas aplicaciones de Windows sin necesidad de Windows.
SCREENSHOTS
Ad
gsasnp2
DESCRIPCIÓN
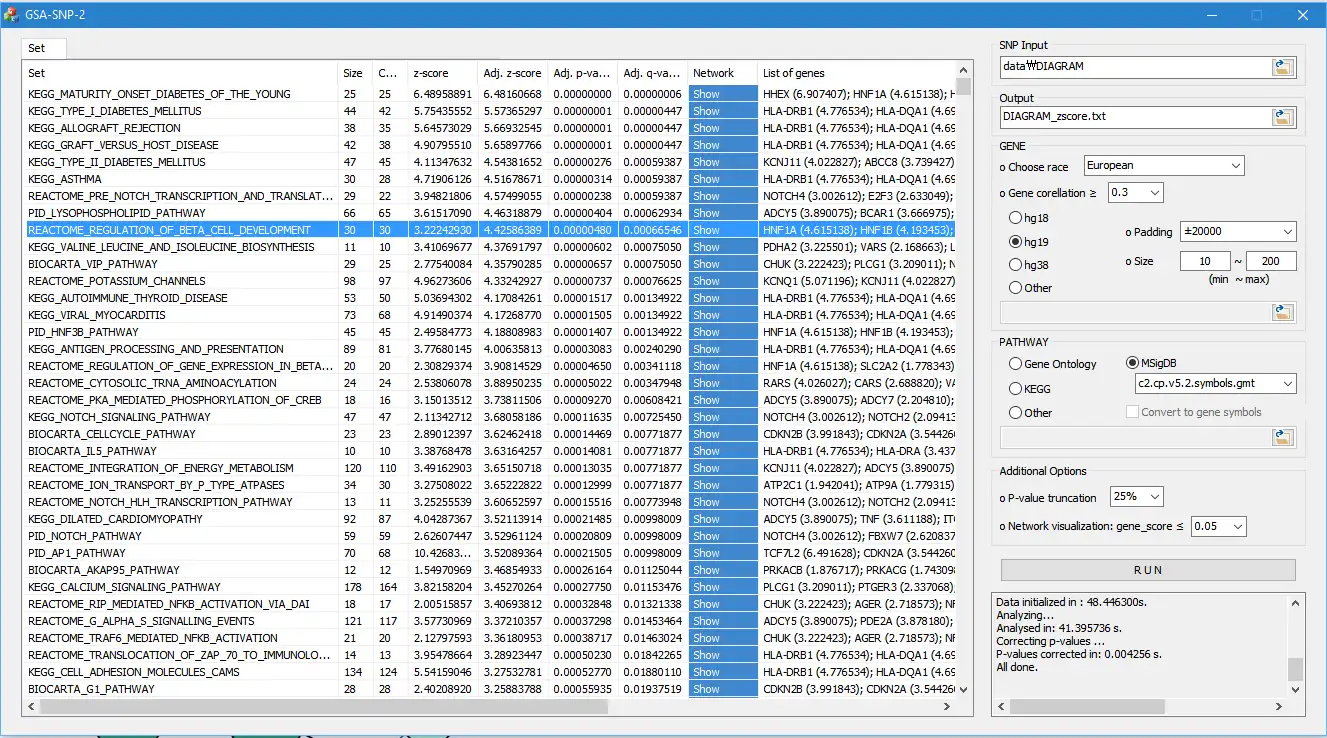
* GSA-SNP2 es un sucesor de GSA-SNP (Nam et al. 2010, problema del servidor web NAR). GSA-SNP2 acepta datos de resumen de GWAS humanos (números rs, valores p) o valores p genéticos y genera conjuntos de genes de la vía 'enriquecidos' con genes asociados con el fenotipo dado. También proporciona redes de interacción de proteínas tanto locales como globales en las vías asociadas.
* Artículo: SYoon, HCTNguyen, YJYoo, JKim, BBaik, SKim, JKim, SKim, DNam, "Enriquecimiento eficiente de vías y análisis de redes de datos resumidos de GWAS usando GSA-SNP2", Investigación de ácidos nucleicos, vol. 46 (10), e60 (2018).
* ID de PubMed: 29562348
* DOI: 10.1093 / nar / gky175
-> POR FAVOR, MUEVA O HAGA UNA COPIA DE LA CARPETA DE 'DATOS' EN SU CARPETA DE PRUEBA INTENSIVA (POR EJEMPLO, CARPETA ESPECIFICADA DE LINUX, MAC O WINDOWS) PARA PERMITIR QUE EL PROGRAMA ENCUENTRE LOS DATOS PREDEFINIDOS.
* ACTUALIZAR NOTA:
-> 1 de septiembre de 2020: agregue una actualización para Ubuntu-20.04. Necesitará la biblioteca Boost instalada (sudo apt-get install libboost-all-dev)
-> 7 de marzo de 2018: revisar los términos del encabezado en el archivo de salida
Caracteristicas
- 1 / 'CONTROL DE ERRORES TIPO I DECENTE' logrado mediante los dos procesos siguientes: A) Las puntuaciones de los genes se 'ajustan' al número de SNP asignados a cada gen utilizando una curva de tendencia spline cúbica monótona. B) Se eliminaron los genes adyacentes con altas correlaciones entre genes dentro de cada vía.
- 2 / 'ALTA POTENCIA Y COMPUTACIÓN RÁPIDA' basado en el modelo de conjunto aleatorio
- 3 / 'SIN PARÁMETRO LIBRE CRÍTICO'
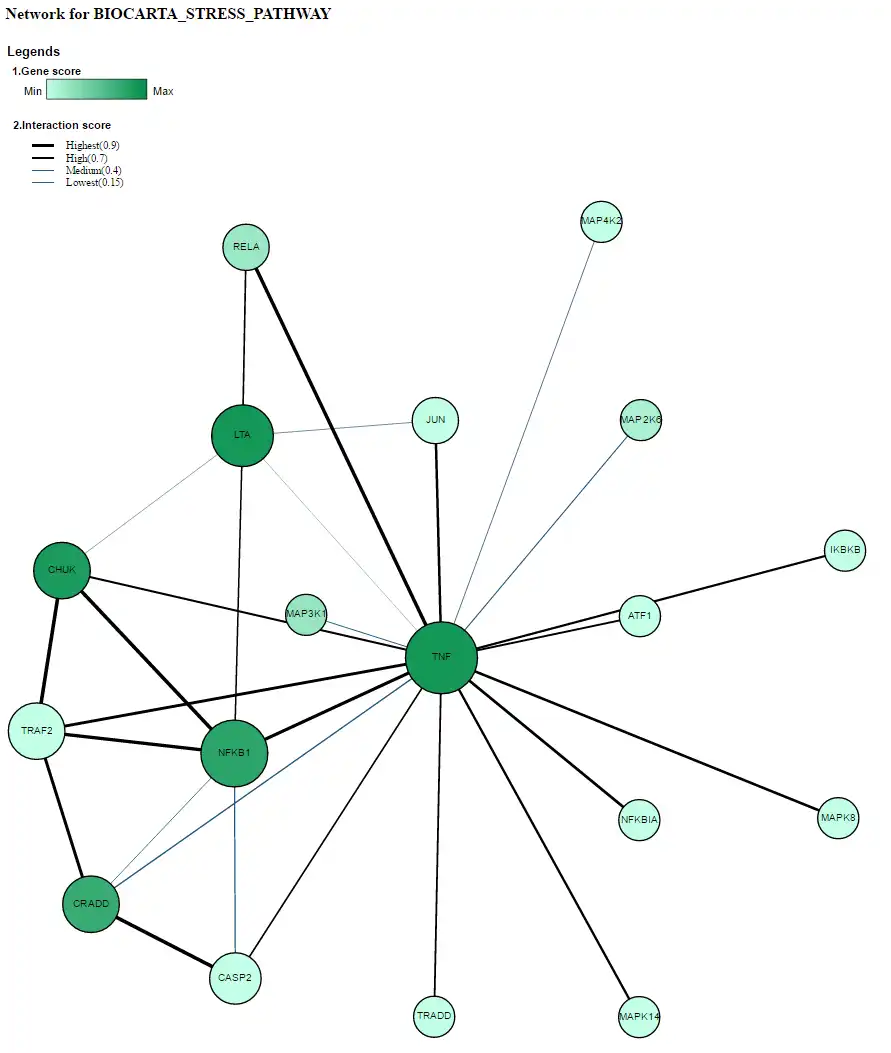
- Se visualizaron 4 / 'REDES DE INTERACCIÓN DE PROTEÍNAS' entre los genes miembros para las vías significativas. Esta función permite al usuario priorizar las subredes centrales dentro y a través de las rutas importantes. Actualmente se proporcionan las redes STRING e HIPPIE
- 5 / 'FÁCIL DE USAR': Solo requiere datos resumidos de GWAS (o valores p de genes) y solo toma uno o dos minutos obtener resultados. Otras potentes herramientas de ruta autónomas también requieren la entrada de correlación SNP y requieren mucho más tiempo. El usuario también puede cargar sus propios conjuntos de genes de vías y redes de interacción de proteínas.
Interfaz de usuario
Win32 (MS Windows), línea de comandos
Lenguaje de programación
C ++, PHP, JavaScript
Categorías
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/gsasnp2/. Se ha alojado en OnWorks para poder ejecutarlo online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.