Esta es la aplicación de Windows llamada Visualization of Protein-Ligand Graphs para ejecutar en Windows en línea sobre Linux en línea, cuya última versión se puede descargar como vplg_2016_07_18.zip. Se puede ejecutar en línea en el proveedor de alojamiento gratuito OnWorks para estaciones de trabajo.
Descargue y ejecute en línea esta aplicación llamada Visualización de gráficos de ligandos de proteínas para ejecutar en Windows en línea sobre Linux en línea con OnWorks de forma gratuita.
Siga estas instrucciones para ejecutar esta aplicación:
- 1. Descargue esta aplicación en su PC.
- 2. Ingrese en nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 3. Cargue esta aplicación en dicho administrador de archivos.
- 4. Inicie cualquier emulador en línea de OS OnWorks desde este sitio web, pero mejor emulador en línea de Windows.
- 5. Desde el sistema operativo OnWorks Windows que acaba de iniciar, vaya a nuestro administrador de archivos https://www.onworks.net/myfiles.php?username=XXXXX con el nombre de usuario que desee.
- 6. Descarga la aplicación e instálala.
- 7. Descargue Wine desde los repositorios de software de sus distribuciones de Linux. Una vez instalada, puede hacer doble clic en la aplicación para ejecutarla con Wine. También puedes probar PlayOnLinux, una elegante interfaz sobre Wine que te ayudará a instalar programas y juegos populares de Windows.
Wine es una forma de ejecutar software de Windows en Linux, pero no requiere Windows. Wine es una capa de compatibilidad de Windows de código abierto que puede ejecutar programas de Windows directamente en cualquier escritorio de Linux. Esencialmente, Wine está tratando de volver a implementar una cantidad suficiente de Windows desde cero para poder ejecutar todas esas aplicaciones de Windows sin necesidad de Windows.
SCREENSHOTS
Ad
Visualización de gráficos de proteína-ligando para ejecutar en Windows en línea sobre Linux en línea
DESCRIPCIÓN
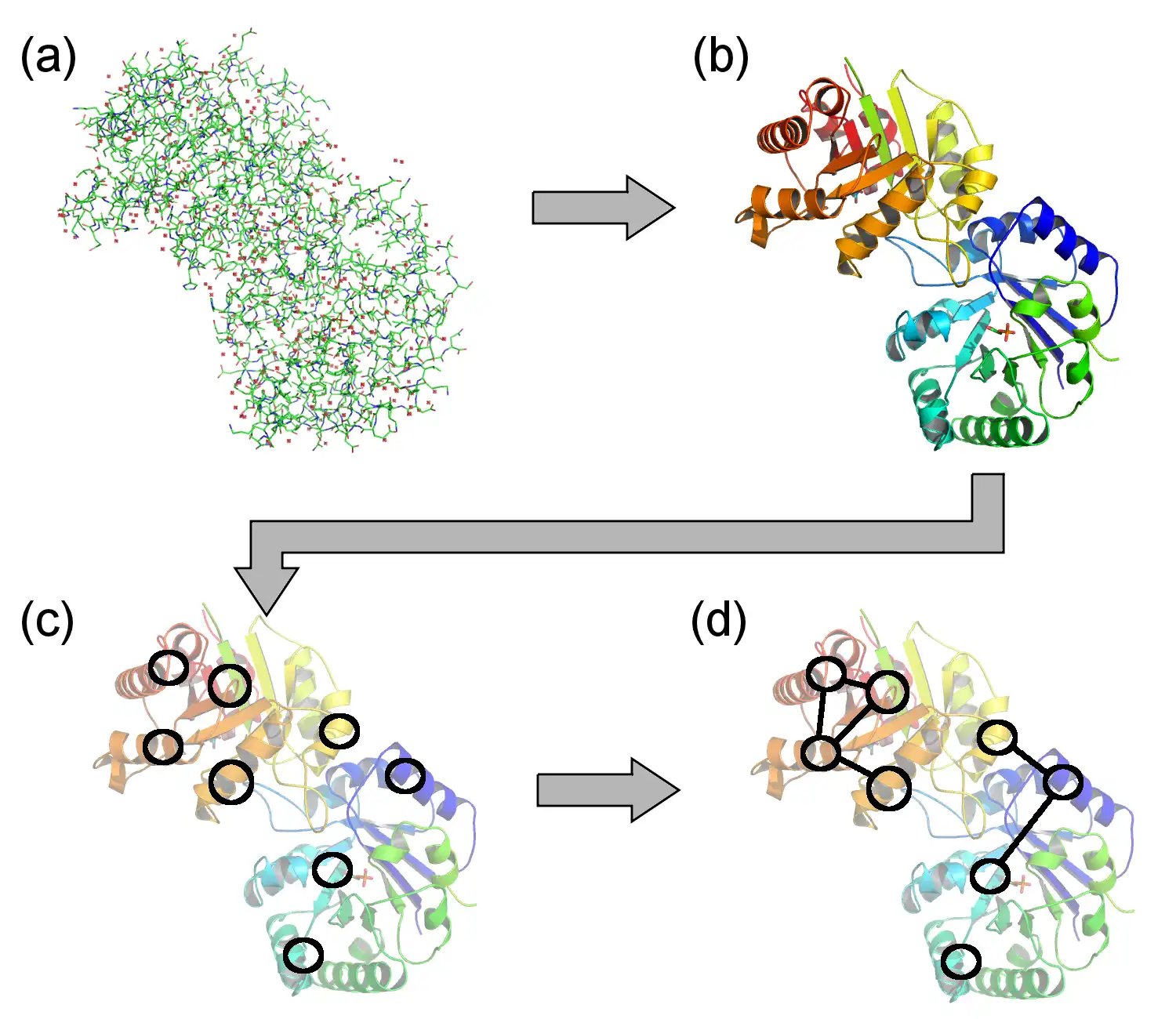
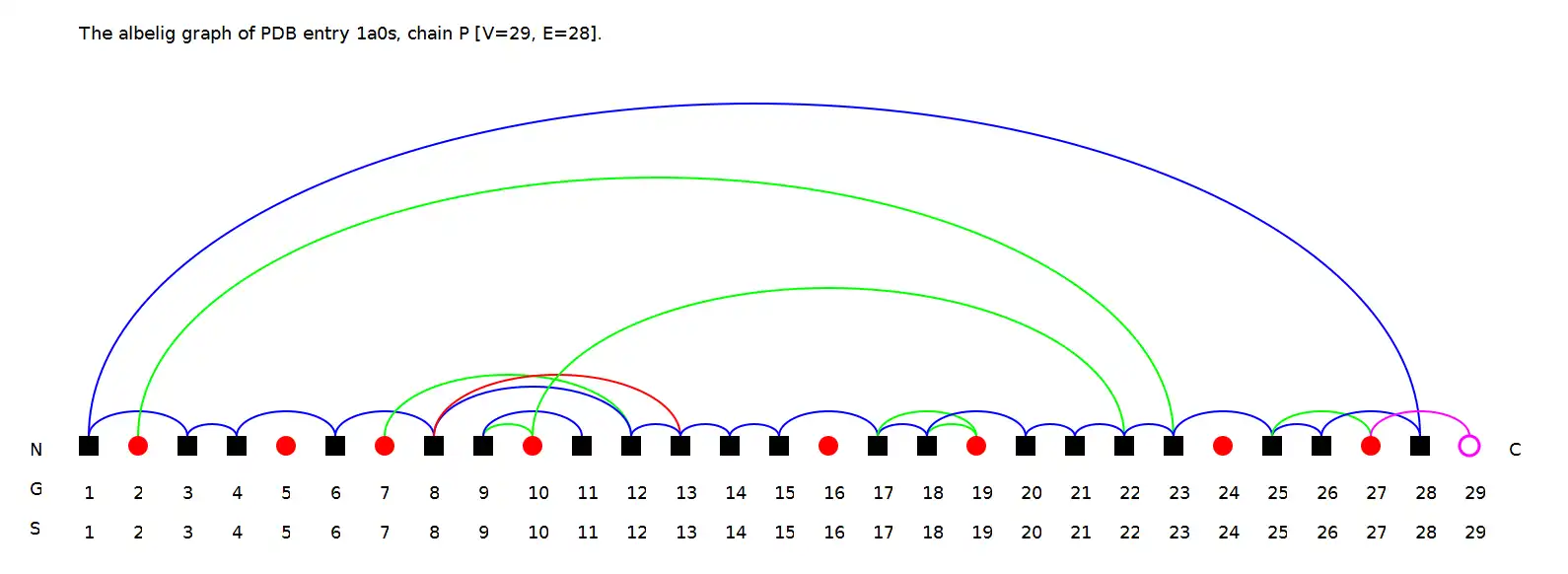
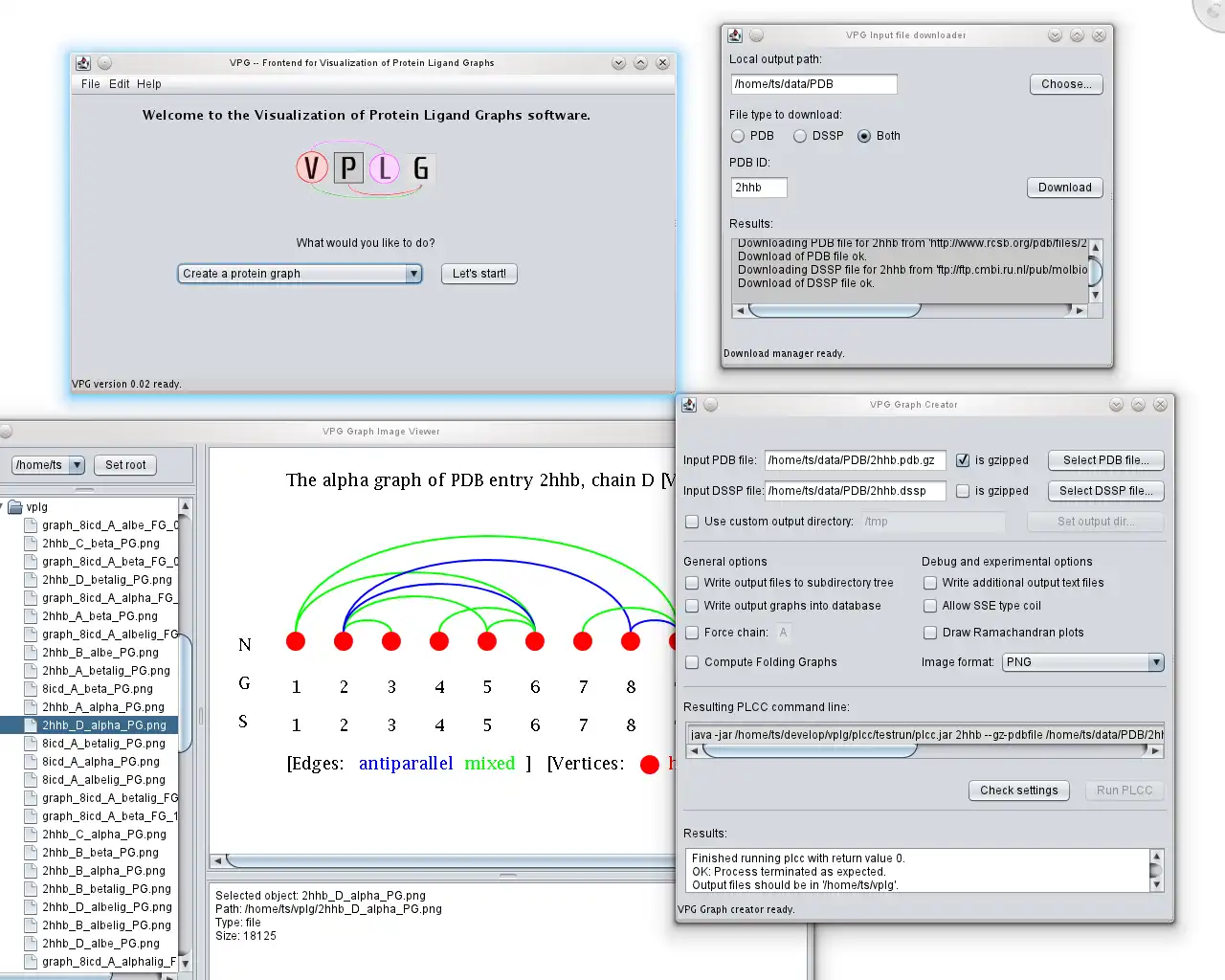
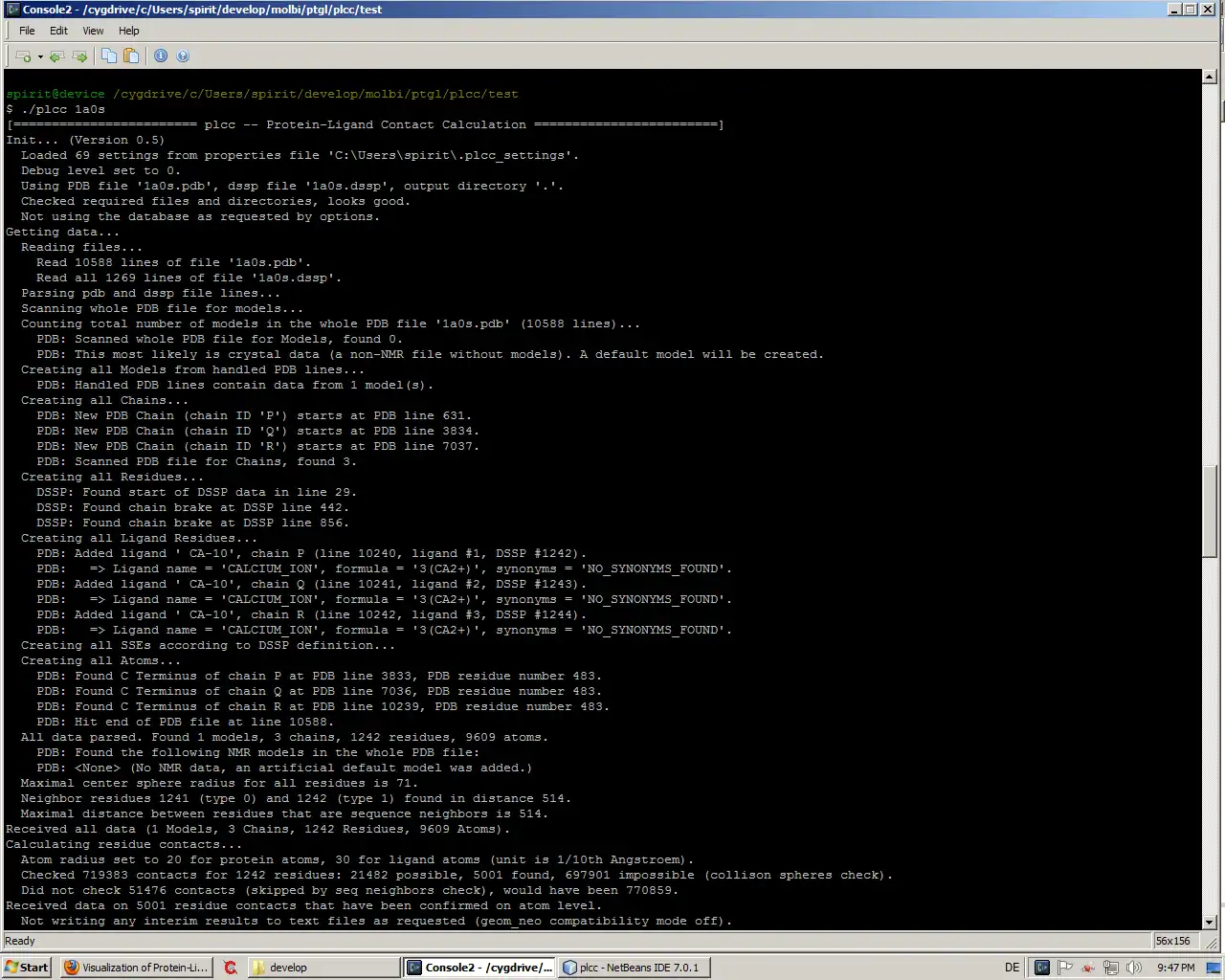
El paquete de software Visualization of Protein-Ligand Graphs (VPLG) calcula y visualiza gráficos de proteínas. Funciona en el nivel de estructura supersecundaria y utiliza las coordenadas del átomo de los archivos PDB y las asignaciones SSE del algoritmo DSSP.VPLG es un software de línea de comandos. Si no le gusta escribir comandos, pruebe nuestro servidor web PTGL: http://ptgl.uni-frankfurt.de/
Caracteristicas
- Lee datos de átomos tridimensionales de archivos PDB y asignaciones de estructuras secundarias de archivos DSSP
- Calcula un gráfico de proteína-ligando a partir de los datos y visualiza el gráfico
- Admite la salida de imágenes de gráficos en formatos de mapa de bits (.png) y vectoriales (.svg)
- Exporta gráficos de proteína-ligando en un archivo de texto sin formato en su propio formato (.plg) que es fácil de editar, analizar y generar con un programa de computadora, así como en varios formatos de archivo de gráfico estándar.
- Puede leer y visualizar directamente gráficos de proteínas de archivos .plg
- Software gratuito de código abierto (FOSS)
- Soporte de base de datos (completamente opcional, no se requiere servidor de base de datos por defecto)
- La interfaz de línea de comandos permite un fácil procesamiento por lotes a través de scripts de shell
- Permite el filtrado de ligandos por número de átomo.
- Nueva GUI escrita en Java, ¡ya no es necesario escribir comandos de consola!
- El software que alimenta el servidor web PTGL: http://ptgl.uni-frankfurt.de/
Audiencia
Ciencia / Investigación, Educación, Usuarios finales avanzados
Interfaz de usuario
Java Swing, consola / terminal, línea de comandos
Lenguaje de programación
Java
Entorno de base de datos
JDBC
Esta es una aplicación que también se puede obtener de https://sourceforge.net/projects/vplg/. Se ha alojado en OnWorks para poder ejecutarse online de la forma más sencilla desde uno de nuestros Sistemas Operativos gratuitos.