این برنامه لینوکس به نام gsasnp2 برای اجرا در لینوکس به صورت آنلاین است که آخرین نسخه آن را می توان با عنوان gsasnp2-linux-cmd-ubuntu.zip دانلود کرد. می توان آن را به صورت آنلاین در ارائه دهنده میزبانی رایگان OnWorks برای ایستگاه های کاری اجرا کرد.
این برنامه با نام gsasnp2 را به صورت آنلاین دانلود و اجرا کنید تا به صورت رایگان در لینوکس با OnWorks اجرا شود.
برای اجرای این برنامه این دستورالعمل ها را دنبال کنید:
- 1. این برنامه را در رایانه شخصی خود دانلود کنید.
- 2. در فایل منیجر ما https://www.onworks.net/myfiles.php?username=XXXXX نام کاربری مورد نظر خود را وارد کنید.
- 3. این برنامه را در چنین فایل منیجر آپلود کنید.
- 4. OnWorks Linux آنلاین یا شبیه ساز آنلاین ویندوز یا شبیه ساز آنلاین MACOS را از این وب سایت راه اندازی کنید.
- 5. از سیستم عامل لینوکس OnWorks که به تازگی راه اندازی کرده اید، به مدیر فایل ما https://www.onworks.net/myfiles.php?username=XXXXX با نام کاربری که می خواهید بروید.
- 6. اپلیکیشن را دانلود کرده، نصب و اجرا کنید.
عکس ها
Ad
gsasnp2 برای اجرای آنلاین در لینوکس
شرح
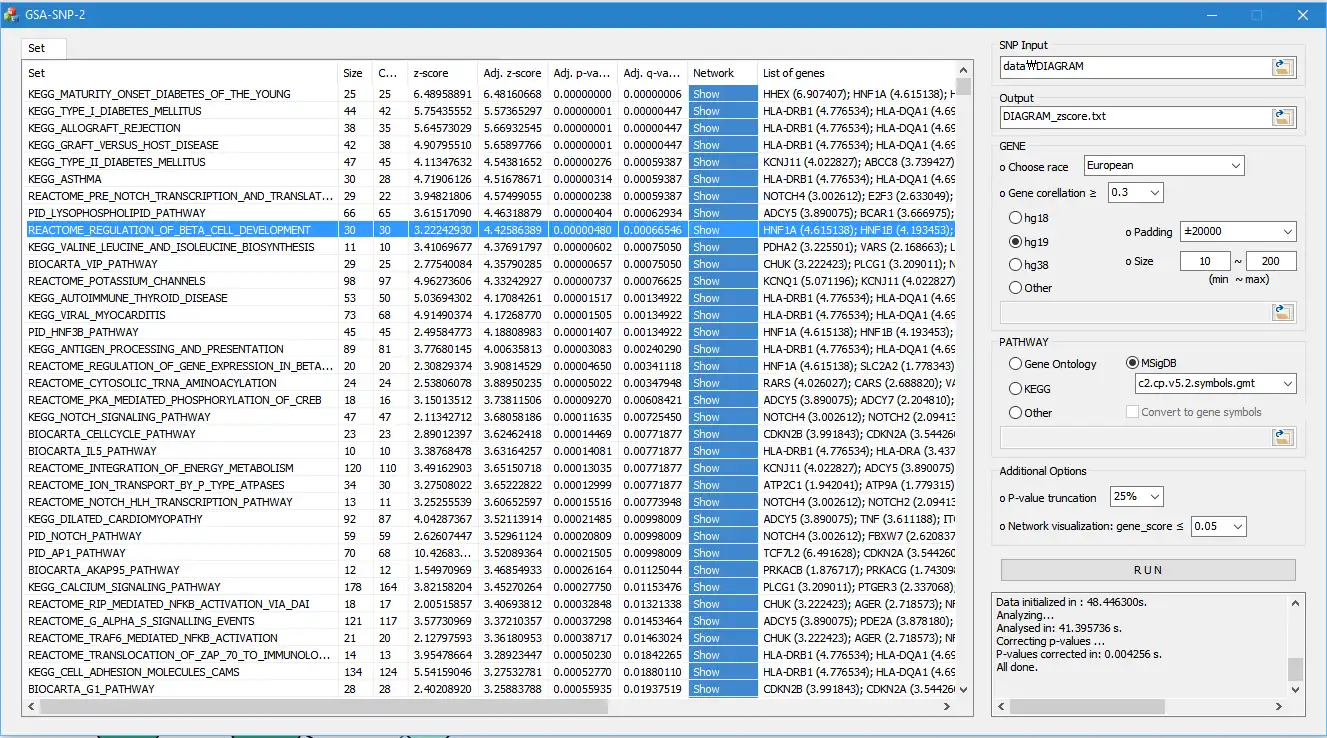
* GSA-SNP2 جانشین GSA-SNP است (نام و همکاران 2010، مسئله سرور وب NAR). GSA-SNP2 دادههای خلاصه GWAS انسانی (اعداد rs، p-value) یا مقادیر p از نظر ژن را میپذیرد و ژنهای مسیر را «غنیشده» با ژنهای مرتبط با فنوتیپ داده شده به بیرون میدهد. همچنین شبکههای تعامل محلی و جهانی پروتئین را در مسیرهای مرتبط فراهم میکند.* مقاله: SYoon، HCTNguyen، YJYoo، JKim، BBaik، SKim، JKim، SKim، DNam، "غنی سازی مسیر کارآمد و تجزیه و تحلیل شبکه داده های خلاصه GWAS با استفاده از GSA-SNP2"، تحقیقات اسیدهای نوکلئیک، جلد. 46 (10)، e60 (2018).
* شناسه PubMed: 29562348
* DOI: 10.1093/nar/gky175
-> لطفاً یک کپی از پوشه "داده" را به پوشه تست فشرده خود (یعنی پوشه مشخص شده در لینوکس، مک یا ویندوز) منتقل کنید یا یک کپی تهیه کنید تا به برنامه اجازه دهید داده های از پیش طراحی شده را پیدا کند.
* توجه به روز رسانی:
-> Aug-7-2019: به روز رسانی برای Ubuntu-19.04 اضافه کنید. شما باید کتابخانه Boost را نصب کنید (sudo apt-get install libboost-all-dev)
-> Mar-7-2018: شرایط هدر را در فایل خروجی اصلاح کنید
امکانات
- 1/ "کنترل خطای نوع I مناسب" با دو فرآیند زیر به دست می آید: الف) امتیازات ژن با تعداد SNP های اختصاص داده شده به هر ژن با استفاده از منحنی روند یکنواخت مکعبی اسپلاین "تعدیل" می شود. ب) ژن های مجاور با همبستگی بین ژنی بالا در هر مسیر حذف شدند
- 2/ «محاسبات پرقدرت و سریع» بر اساس مدل مجموعه تصادفی
- 3/ "بدون پارامتر رایگان حیاتی"
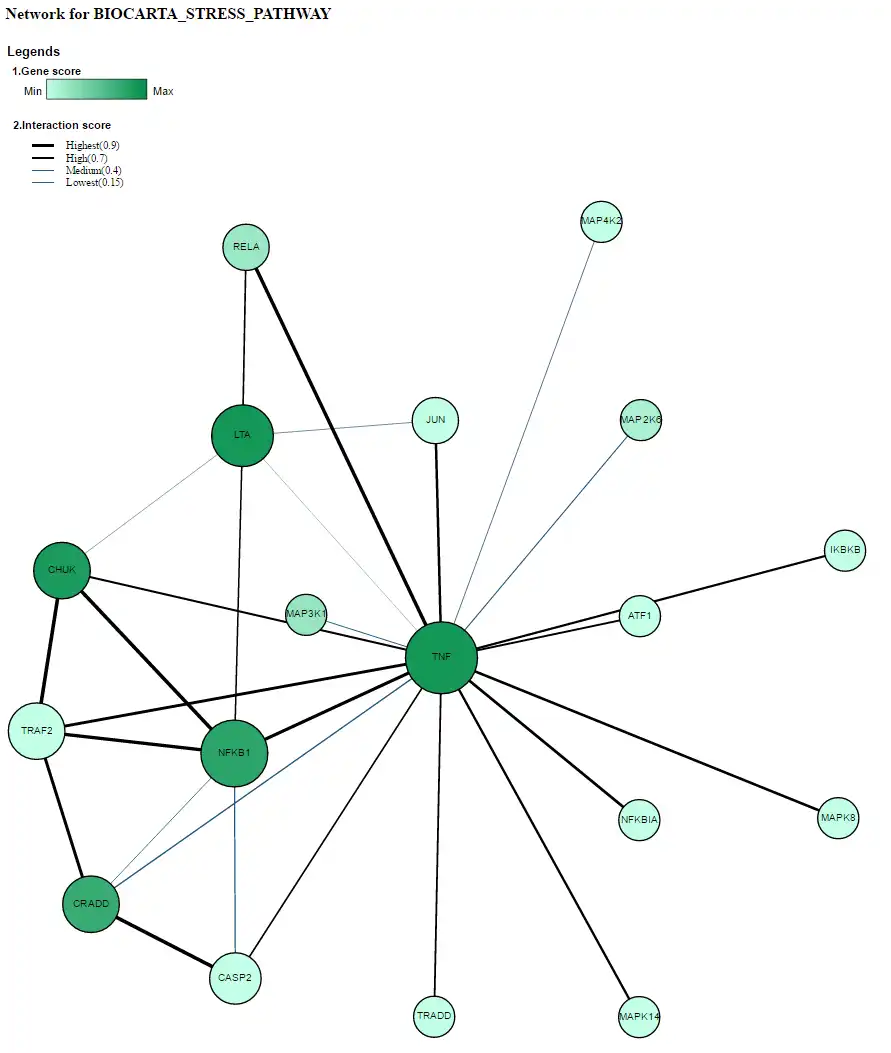
- 4/ "شبکه های برهمکنش پروتئین" در بین ژن های عضو برای مسیرهای قابل توجه مشاهده شد. این تابع کاربر را قادر می سازد تا زیرشبکه های اصلی را در داخل و در مسیرهای مهم اولویت بندی کند. شبکه های STRING و HIPPIE در حال حاضر ارائه شده اند
- 5/ "استفاده آسان": فقط به داده های خلاصه GWAS (یا مقادیر p ژن) نیاز دارد و فقط یک یا دو دقیقه طول می کشد تا به نتیجه برسد. سایر ابزارهای قدرتمند مسیر مستقل به ورودی همبستگی SNP نیز نیاز دارند و زمان بسیار بیشتری را صرف می کنند. کاربر همچنین میتواند مجموعههای ژنی مسیر و شبکههای تعامل پروتئینی خود را آپلود کند.
رابط کاربری
Win32 (MS Windows)، خط فرمان
زبان برنامه نویسی
C++، PHP، JavaScript
این برنامه ای است که می توان آن را از https://sourceforge.net/projects/gsasnp2/ نیز دریافت کرد. در OnWorks میزبانی شده است تا به آسانی از یکی از سیستم عامل های رایگان ما به صورت آنلاین اجرا شود.