Il s'agit de l'application Windows nommée Comparative synteny analysis viewer dont la dernière version peut être téléchargée sous comparative_synteny.zip. Il peut être exécuté en ligne dans le fournisseur d'hébergement gratuit OnWorks pour les postes de travail.
Téléchargez et exécutez en ligne cette application nommée Visionneuse d'analyse comparative de la synténie avec OnWorks gratuitement.
Suivez ces instructions pour exécuter cette application :
- 1. Téléchargé cette application sur votre PC.
- 2. Entrez dans notre gestionnaire de fichiers https://www.onworks.net/myfiles.php?username=XXXXX avec le nom d'utilisateur que vous voulez.
- 3. Téléchargez cette application dans ce gestionnaire de fichiers.
- 4. Démarrez n'importe quel émulateur en ligne OS OnWorks à partir de ce site Web, mais un meilleur émulateur en ligne Windows.
- 5. Depuis le système d'exploitation OnWorks Windows que vous venez de démarrer, accédez à notre gestionnaire de fichiers https://www.onworks.net/myfiles.php?username=XXXXX avec le nom d'utilisateur que vous souhaitez.
- 6. Téléchargez l'application et installez-la.
- 7. Téléchargez Wine depuis les dépôts de logiciels de vos distributions Linux. Une fois installé, vous pouvez ensuite double-cliquer sur l'application pour les exécuter avec Wine. Vous pouvez également essayer PlayOnLinux, une interface sophistiquée sur Wine qui vous aidera à installer des programmes et des jeux Windows populaires.
Wine est un moyen d'exécuter un logiciel Windows sur Linux, mais sans Windows requis. Wine est une couche de compatibilité Windows open source qui peut exécuter des programmes Windows directement sur n'importe quel bureau Linux. Essentiellement, Wine essaie de ré-implémenter suffisamment de Windows à partir de zéro pour qu'il puisse exécuter toutes ces applications Windows sans avoir réellement besoin de Windows.
CAPTURES D'ÉCRAN
Ad
Visionneuse d'analyse de synténie comparative
DESCRIPTION
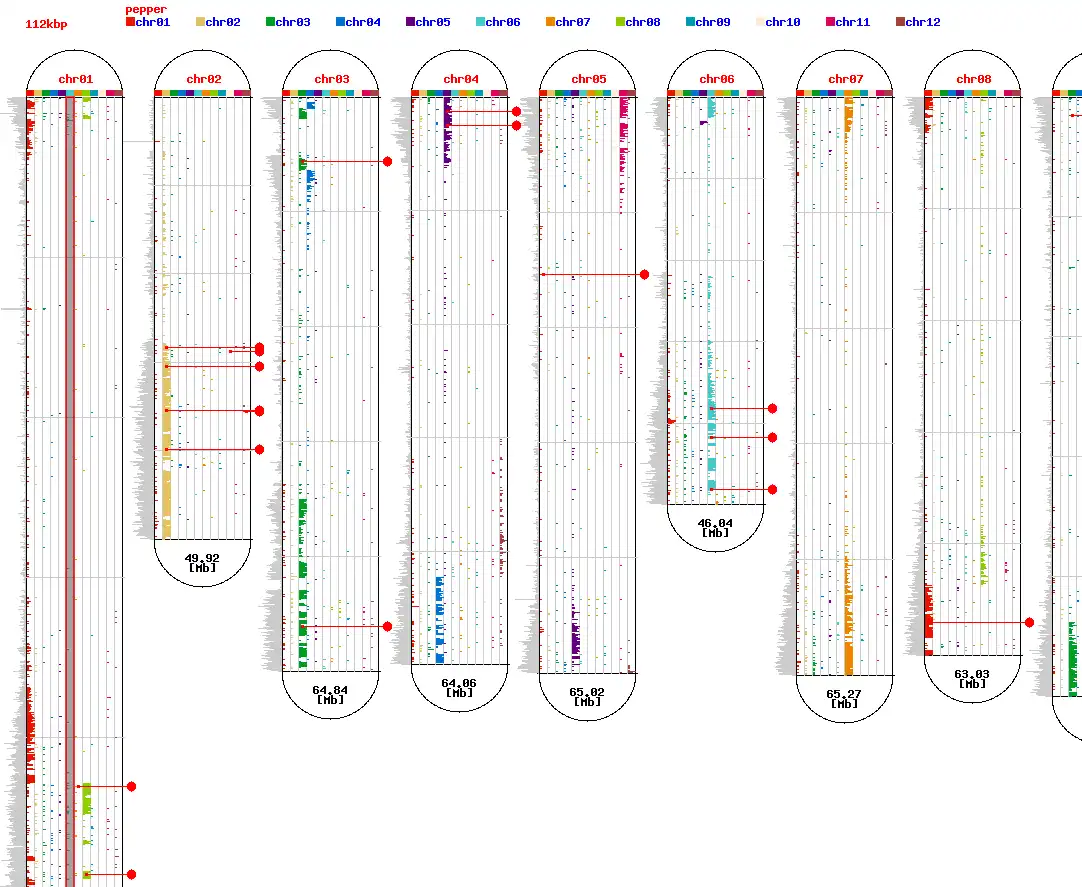
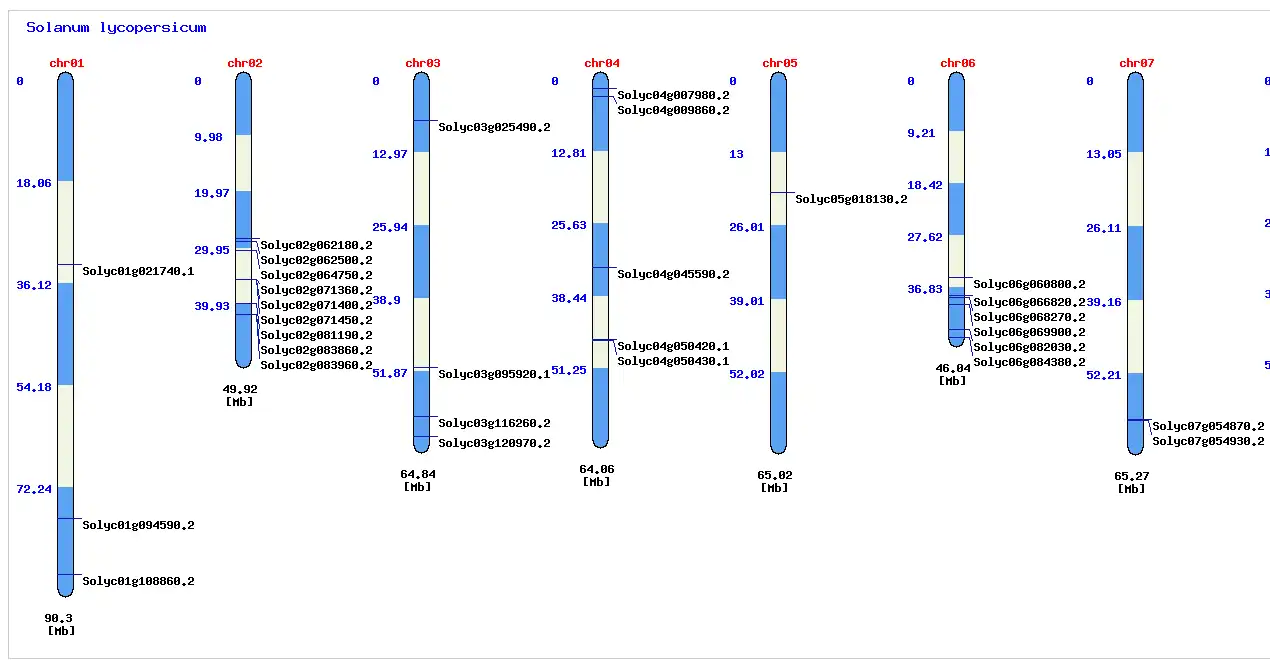
Un outil d'analyse comparative de la synténie pour la découverte de marqueurs SNP du gène cible : connecter les données génomiques à la sélection chez les solanacées* La base de cette source Web est CodeIgniter.
* Après avoir défini le framework CodeIgniter, décompressez-le dans le répertoire personnel par défaut.
La base de données est basée sur Maria DB (mysql).
Téléchargez le schéma et importez-le dans la base de données.
Chaque espèce que vous souhaitez analyser a besoin de résultats explosifs.
Ajustez les champs pour correspondre au schéma et cela fonctionnera.
Il s'agit d'une application qui peut également être récupérée à partir de https://sourceforge.net/projects/seederscs/. Il a été hébergé dans OnWorks afin d'être exécuté en ligne de la manière la plus simple à partir de l'un de nos systèmes d'exploitation gratuits.