Il s'agit de l'application Windows nommée NGSEP à exécuter sous Windows en ligne sur Linux en ligne dont la dernière version peut être téléchargée en tant que NGSEPcore_4.0.1.jar. Il peut être exécuté en ligne sur le fournisseur d'hébergement gratuit OnWorks pour les postes de travail.
Téléchargez et exécutez en ligne cette application nommée NGSEP pour une exécution gratuite sous Windows en ligne sur Linux en ligne avec OnWorks.
Suivez ces instructions pour exécuter cette application :
- 1. Téléchargé cette application sur votre PC.
- 2. Entrez dans notre gestionnaire de fichiers https://www.onworks.net/myfiles.php?username=XXXXX avec le nom d'utilisateur que vous voulez.
- 3. Téléchargez cette application dans ce gestionnaire de fichiers.
- 4. Démarrez n'importe quel émulateur en ligne OS OnWorks à partir de ce site Web, mais un meilleur émulateur en ligne Windows.
- 5. Depuis le système d'exploitation OnWorks Windows que vous venez de démarrer, accédez à notre gestionnaire de fichiers https://www.onworks.net/myfiles.php?username=XXXXX avec le nom d'utilisateur que vous souhaitez.
- 6. Téléchargez l'application et installez-la.
- 7. Téléchargez Wine depuis les dépôts de logiciels de vos distributions Linux. Une fois installé, vous pouvez ensuite double-cliquer sur l'application pour les exécuter avec Wine. Vous pouvez également essayer PlayOnLinux, une interface sophistiquée sur Wine qui vous aidera à installer des programmes et des jeux Windows populaires.
Wine est un moyen d'exécuter un logiciel Windows sur Linux, mais sans Windows requis. Wine est une couche de compatibilité Windows open source qui peut exécuter des programmes Windows directement sur n'importe quel bureau Linux. Essentiellement, Wine essaie de ré-implémenter suffisamment de Windows à partir de zéro pour qu'il puisse exécuter toutes ces applications Windows sans avoir réellement besoin de Windows.
CAPTURES D'ÉCRAN
Ad
NGSEP pour fonctionner sous Windows en ligne sur Linux en ligne
DESCRIPTION
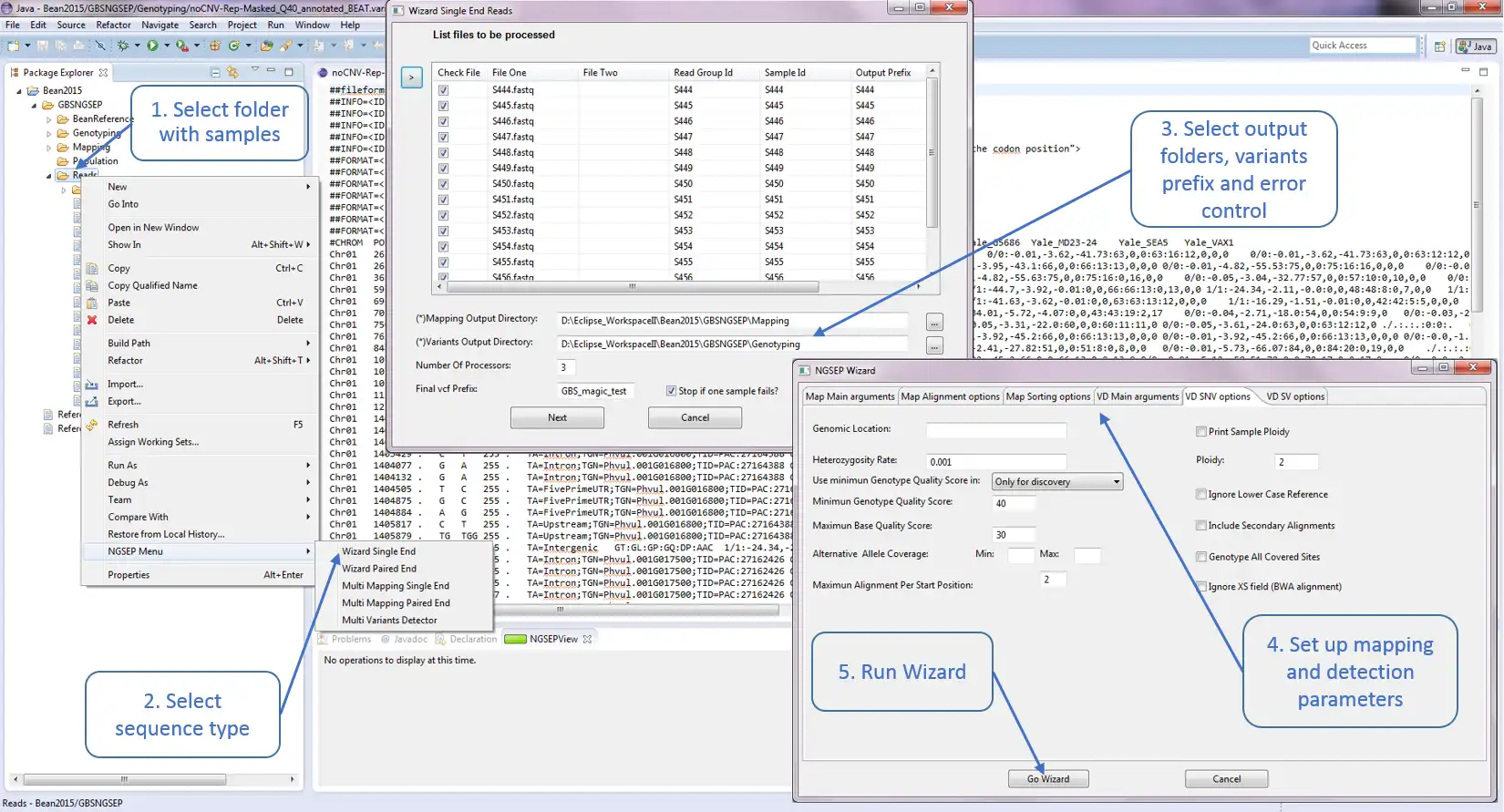
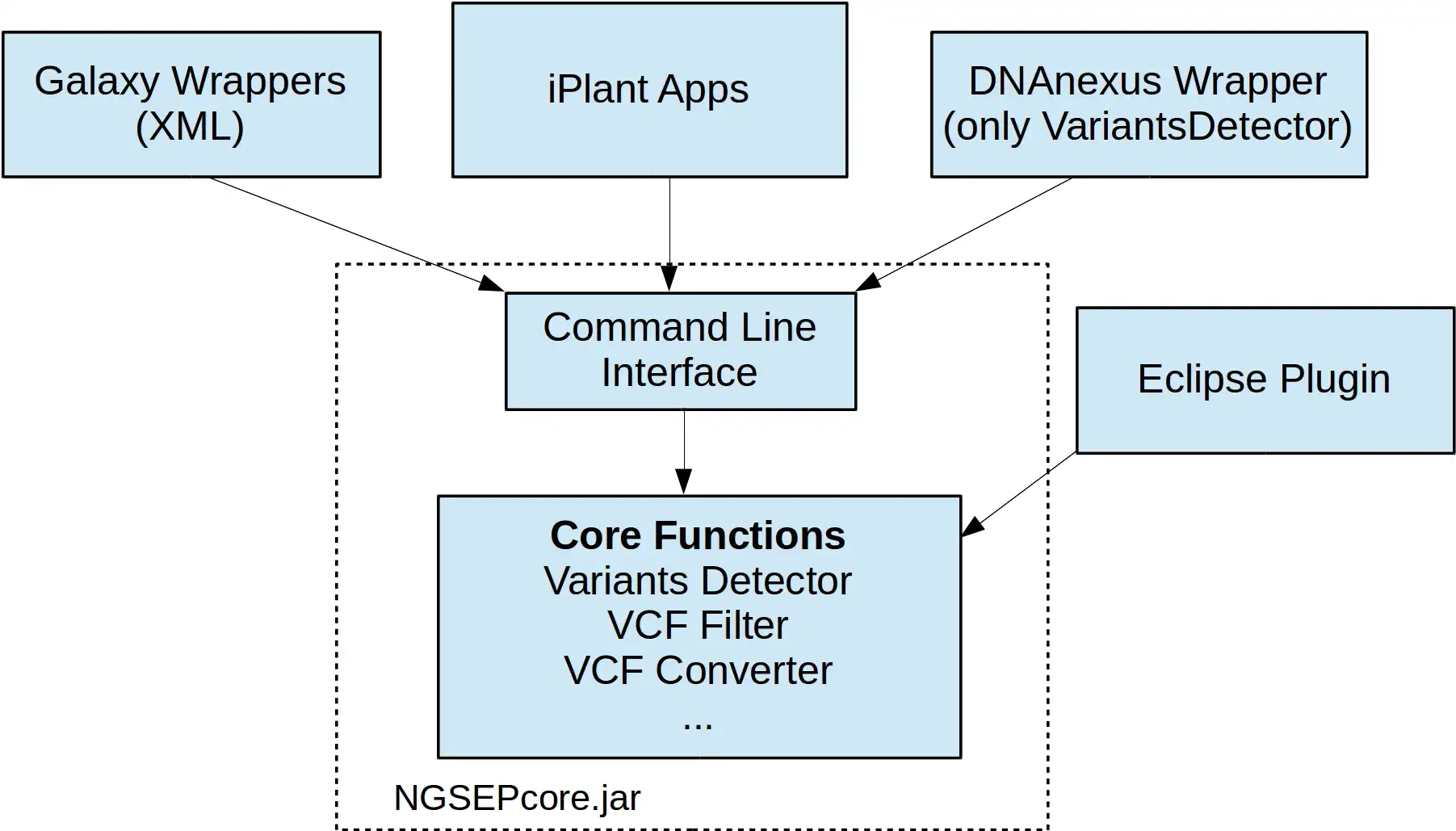
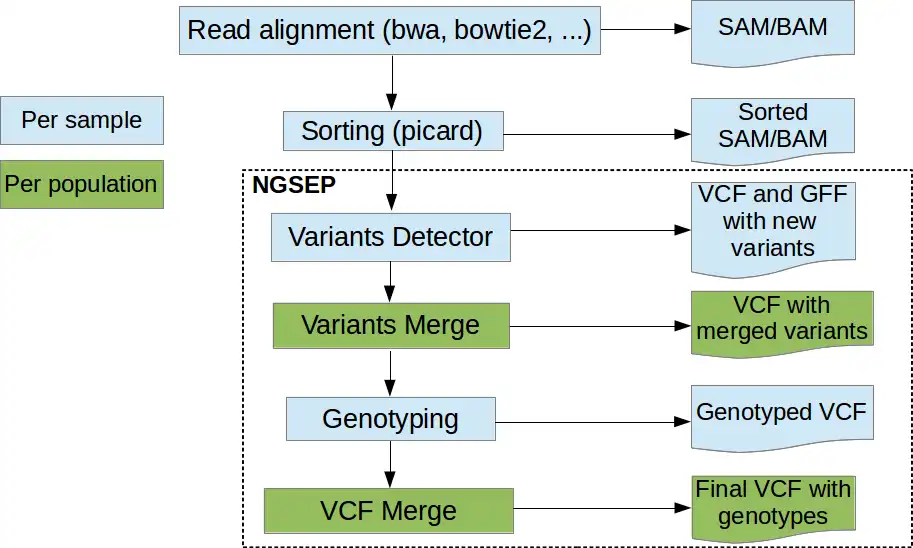
NGSEP est un cadre intégré pour l'analyse des données de séquençage d'ADN à haut débit. L'utilisation principale du NGSEP est la construction et l'analyse en aval de grands ensembles de données de variation génomique. NGSEP effectue une détection et un génotypage précis des variants nucléotidiques simples (SNV), des indels petits et grands, des répétitions en tandem courtes (STR), des inversions et des variants du nombre de copies (CNV). NGSEP fournit également des modules pour l'annotation fonctionnelle, le filtrage, la conversion de format, la comparaison, le regroupement, l'imputation, l'analyse d'introgression et différents types de statistiques. Une liste complète des fonctionnalités est disponible dans notre wiki (https://sourceforge.net/p/ngsep/wiki/Home/).NEWS : Nous venons de mettre à disposition la première version en ligne de commande de la version 4, y compris notre propre implémentation d'un aligneur de lecture basé sur l'index FM. L'utilisation de la ligne de commande a été standardisée sur l'ensemble de l'application. Veuillez prendre le temps de mettre à jour les scripts pour passer à cette nouvelle version. Plus de détails sont disponibles dans le wiki.
Caractéristiques
- Détection de SNP, CNV et Variantes Structurelles
- Alignement des lectures brutes sur un génome de référence
- Manipulation VCF : annotation fonctionnelle, fusion, filtrage, comparaison, conversion de format, imputation
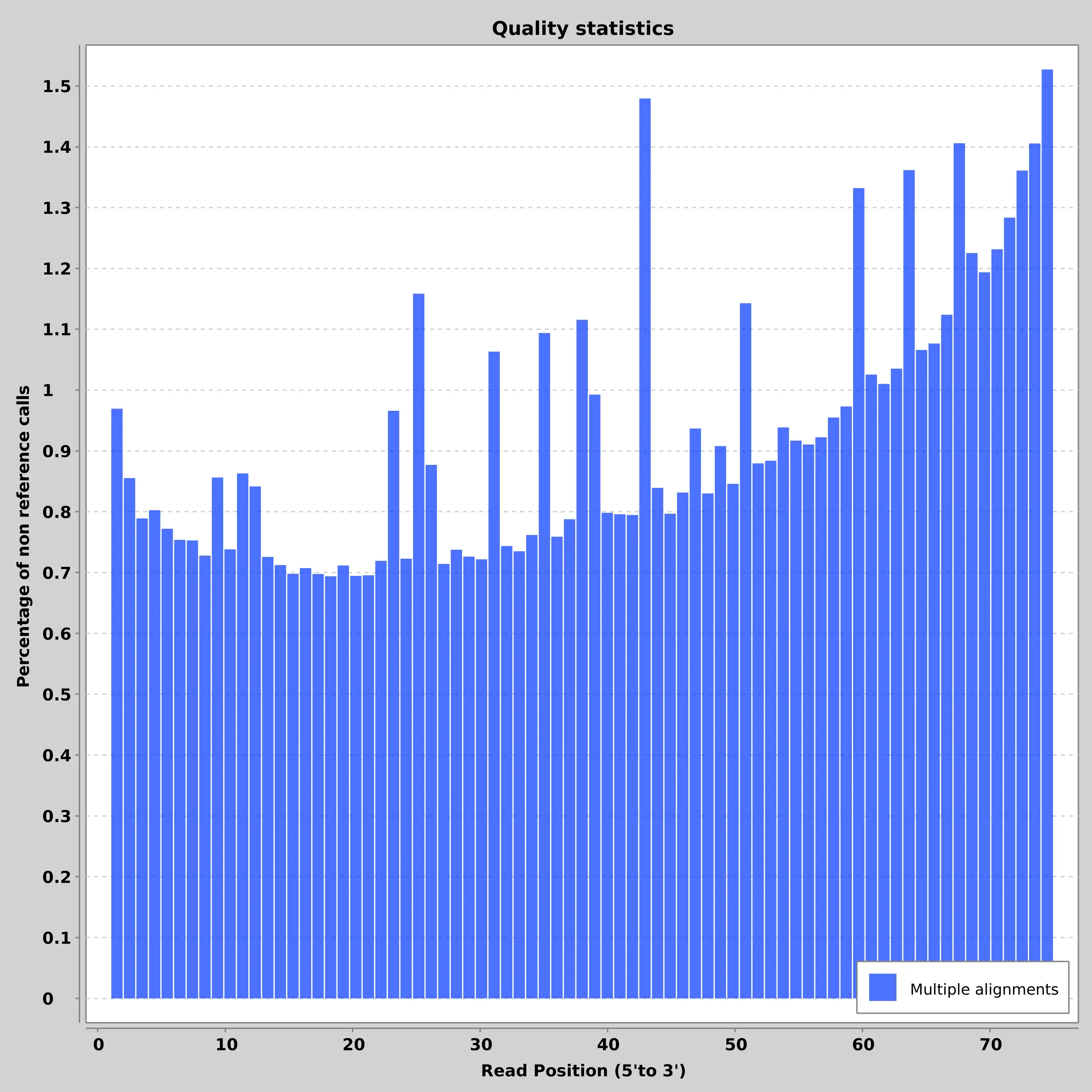
- Calcul et tracé des statistiques SAM/BAM et VCF
- Démultiplexage des lectures
- Alignement des assemblages de génomes annotés
- Statistiques et filtrage sur les annulations du transcriptome au format GFF3
Audience
Industrie de la santé, Science/Recherche, Autre public, Agriculture
Interface utilisateur
Java SWT, console/terminal, Eclipse
Langage de programmation
Java
Il s'agit d'une application qui peut également être récupérée à partir de https://sourceforge.net/projects/ngsep/. Il a été hébergé dans OnWorks afin d'être exécuté en ligne de la manière la plus simple à partir de l'un de nos systèmes d'exploitation gratuits.