This is the Linux app named BIRAP whose latest release can be downloaded as Sample_Files.zip. It can be run online in the free hosting provider OnWorks for workstations.
Download and run online this app named BIRAP with OnWorks for free.
Follow these instructions in order to run this app:
- 1. Downloaded this application in your PC.
- 2. Enter in our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 3. Upload this application in such filemanager.
- 4. Start the OnWorks Linux online or Windows online emulator or MACOS online emulator from this website.
- 5. From the OnWorks Linux OS you have just started, goto our file manager https://www.onworks.net/myfiles.php?username=XXXXX with the username that you want.
- 6. Download the application, install it and run it.
SCREENSHOTS
Ad
BIRAP
DESCRIPTION
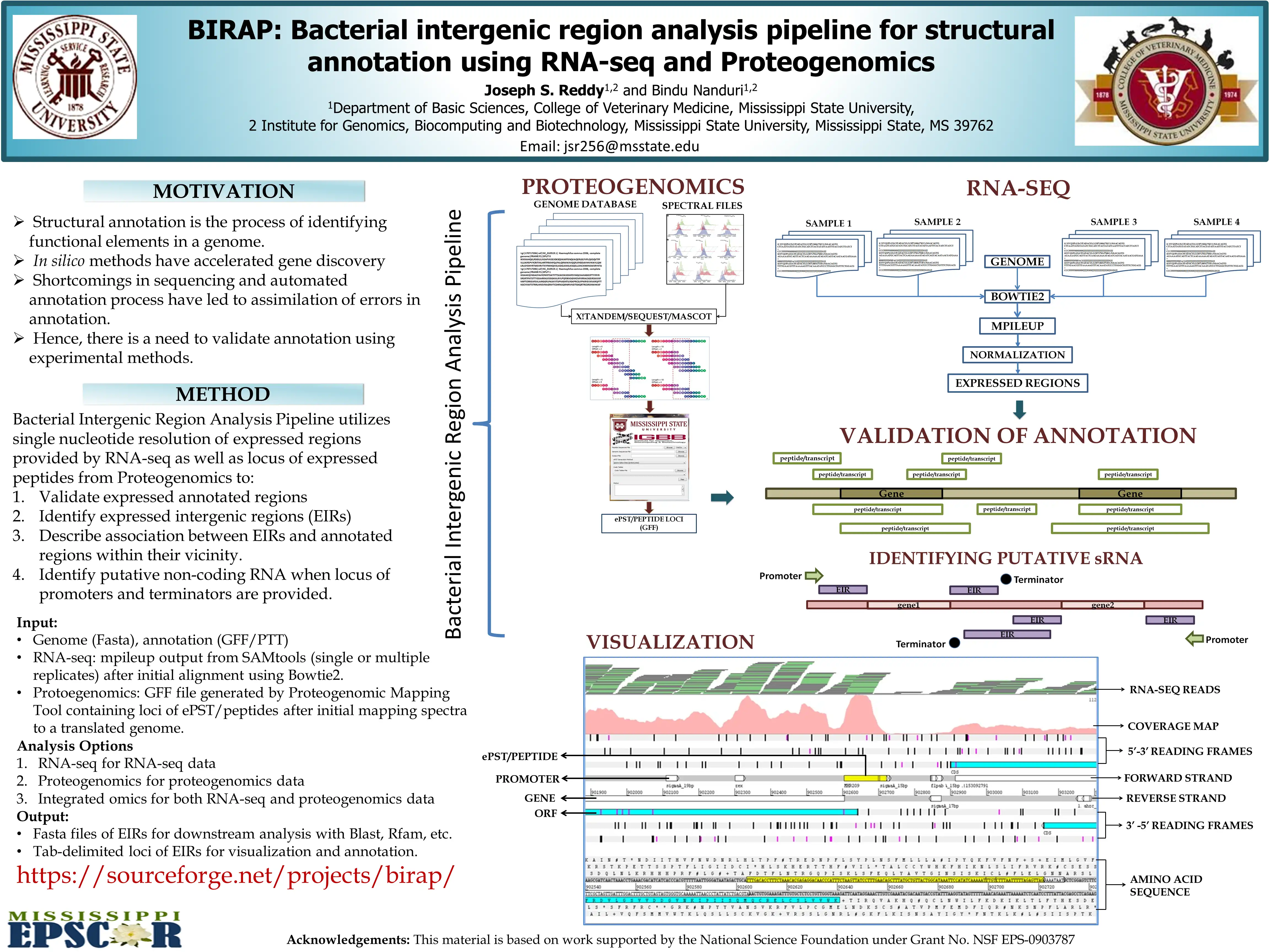
BIRAP (Bacterial Intergenic Region Analysis Pipeline) is an open source, easy to use Perl pipeline that can be used to re-annotate bacterial genomes using experimental data. The tool integrates expression profile derived from RNA-seq and/or proteogenomics, compares it with existing in silico annotation and helps validate annotation, identify novel protein coding regions, putative non-coding RNA as well as help correct er-rors in the existing annotation. The pipeline requires “pileup” output from SAMtools for RNA-seq data and peptide/ePST locus file (GFF) from the Proteogenomic Mapping tool for prote-ogenomics data along with the genome and existing in silico annotation. When information regarding the locus of promot-ers and terminators in the genome (in .coords format) is pro-vided, the pipeline will also help identify non-coding RNA.
Features
- intergenic region analysis of bacterial genomes
- re-annotation of bacterial genomes
- small RNA (sRNA) detection
- RNA-seq
- Proteogenomics
Audience
Science/Research
User interface
Console/Terminal, Command-line
Programming Language
Perl
Categories
This is an application that can also be fetched from https://sourceforge.net/projects/birap/. It has been hosted in OnWorks in order to be run online in an easiest way from one of our free Operative Systems.