זוהי אפליקציית לינוקס בשם RNAseqR שניתן להוריד את המהדורה האחרונה שלה בתור RNAseqR_1.1.tar.gz. ניתן להפעיל אותו באופן מקוון בספק האירוח החינמי OnWorks עבור תחנות עבודה.
הורד והפעל באינטרנט את האפליקציה הזו בשם RNAseqR עם OnWorks בחינם.
בצע את ההוראות הבאות כדי להפעיל את האפליקציה הזו:
- 1. הורד את היישום הזה למחשב שלך.
- 2. הזן במנהל הקבצים שלנו https://www.onworks.net/myfiles.php?username=XXXXX עם שם המשתמש שאתה רוצה.
- 3. העלה את היישום הזה במנהל קבצים כזה.
- 4. הפעל את האמולטור המקוון של OnWorks Linux או Windows מקוון או אמולטור מקוון של MACOS מאתר זה.
- 5. ממערכת ההפעלה OnWorks Linux שזה עתה התחלת, עבור אל מנהל הקבצים שלנו https://www.onworks.net/myfiles.php?username=XXXXX עם שם המשתמש הרצוי.
- 6. הורד את האפליקציה, התקן אותה והפעל אותה.
בצילומי מסך
Ad
RNAseqR
תיאור
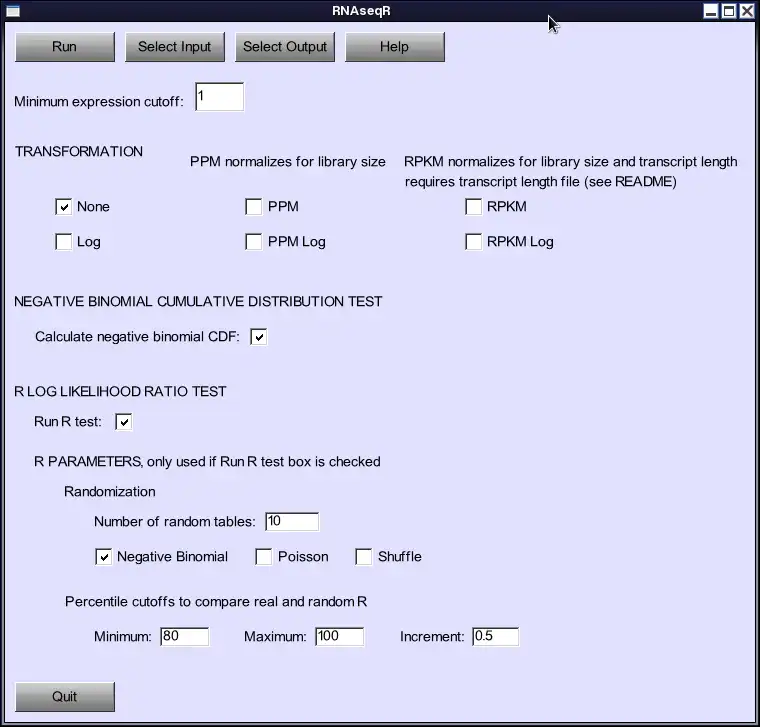
RNAseqR מיועד לניתוח ביטוי של נתוני RNA-seq, והוא המתאים ביותר לניתוח של ספריות מרובות שאינן משוכפלות. זוהי תוכנית מקודדת C++ המורכבת כעת עבור מערכות לינוקס.
עם ממשקי GUI ושורת פקודה, הוא יכול לבצע טרנספורמציות יומן, PPM ו/או RPKM (אם מסופקים אורכים), כמו גם ניתוח סטטיסטי לביטוי דיפרנציאלי, באמצעות פונקציית ההתפלגות המצטברת הבינומית השלילית (CDF) או סטטיסטיקת מבחן R הוצג לניתוח EST על ידי Stekel, et al.
הפלט מאפשר קבלת החלטות על סמך הסתברויות CDF, ערכי R עליונים או השוואה של ערכי R עם נתונים אקראיים. השוואות הן מדד האמינות של Stekel et al, או שנצפה מול R צפוי בביטוי ממוצע נתון. האקראיות היא באמצעות התפלגויות בינומיות שליליות של Poisson, או עירוב עמודות.
שפת תכנות
C + +
כל הקטגוריות
זוהי אפליקציה שניתן להביא גם מ-https://sourceforge.net/projects/rnaseqr/. זה התארח ב-OnWorks על מנת להפעיל אותו באינטרנט בצורה הקלה ביותר מאחת ממערכות ההפעלה החינמיות שלנו.