זוהי אפליקציית לינוקס בשם sgRNAcas9 שניתן להוריד את המהדורה האחרונה שלה בתור sgRNAcas9_3.0.5.zip. ניתן להפעיל אותו באופן מקוון בספק האירוח החינמי OnWorks עבור תחנות עבודה.
הורד והפעל באינטרנט את האפליקציה הזו בשם sgRNAcas9 עם OnWorks בחינם.
בצע את ההוראות הבאות כדי להפעיל את האפליקציה הזו:
- 1. הורד את היישום הזה למחשב שלך.
- 2. הזן במנהל הקבצים שלנו https://www.onworks.net/myfiles.php?username=XXXXX עם שם המשתמש שאתה רוצה.
- 3. העלה את היישום הזה במנהל קבצים כזה.
- 4. הפעל את האמולטור המקוון של OnWorks Linux או Windows מקוון או אמולטור מקוון של MACOS מאתר זה.
- 5. ממערכת ההפעלה OnWorks Linux שזה עתה התחלת, עבור אל מנהל הקבצים שלנו https://www.onworks.net/myfiles.php?username=XXXXX עם שם המשתמש הרצוי.
- 6. הורד את האפליקציה, התקן אותה והפעל אותה.
בצילומי מסך
Ad
sgRNAcas9
תיאור
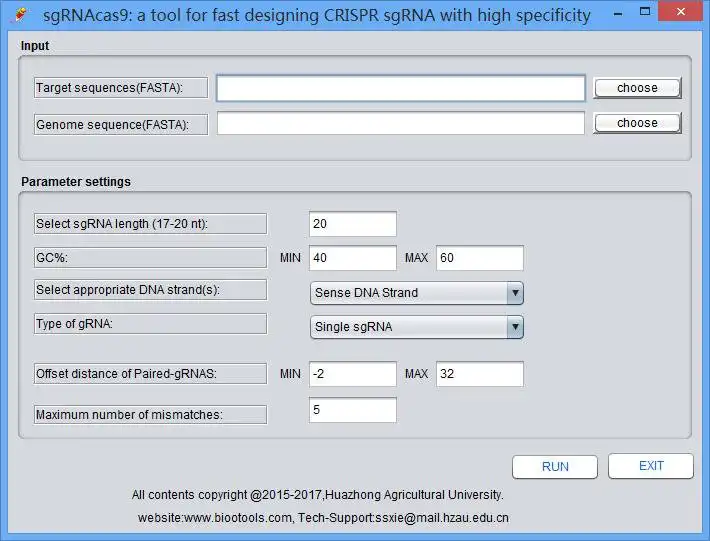
חבילה זו מורכבת מתכניות לביצוע חיפוש של אתרי יעד של CRISPR (פרוטו-ספייסרים) עם פרמטרים המוגדרים על ידי המשתמש, לחזות אתרי מחשוף פוטנציאליים של Cas9 פוטנציאליים מחוץ למטרה (POT), לסווג את ה-POT לשלוש קטגוריות, אוליגונוקלאוטידים לעיצוב אצווה לבניית 20 -nt (נוקלאוטידים) או וקטורי ביטוי של sgRNA קטומים, מחלצים רצפי נוקלאוטידים באורך הרצוי שמאגפים את אתרי המחשוף על או מחוץ למטרה לתכנון צמדי פריימר של PCR לאימות המוטציות על ידי בדיקת חיתוך T7E1. חשוב לציין, על ידי זיהוי אתרים פוטנציאליים מחוץ למטרה בסיליקו, ה-sgRNAcas9 מאפשר בחירה של אתרי יעד ספציפיים יותר ומסייע בזיהוי של אתרים מחוץ למטרה בתום לב, מה שמקל באופן משמעותי על העיצוב של sgRNA עבור יישומי עריכת גנום.מצטט: Xie S, ShenB,Zhang C, Huang X, Zhang Y. sgRNAcas9: חבילת תוכנה לעיצוב CRISPR sgRNA והערכת אתרי מחשוף פוטנציאליים מחוץ למטרה. PLoS ONE. 2014,9(6):e100448.
http://www.biootools.com/
זוהי אפליקציה שניתן להביא גם מ-https://sourceforge.net/projects/sgrnacas9/. הוא התארח ב-OnWorks על מנת להפעיל אותו באינטרנט בצורה הקלה ביותר מאחת ממערכות ההפעלה החינמיות שלנו.