これは MANTI.pl / muda.pl という名前の Linux アプリで、最新リリースは muda.pl-v3.2.zip としてダウンロードできます。 ワークステーション用の無料ホスティング プロバイダー OnWorks でオンラインで実行できます。
MANTI.pl / muda.pl という名前のこのアプリを OnWorks で無料でオンラインでダウンロードして実行します。
このアプリを実行するには、次の手順に従ってください。
-1。このアプリケーションをPCにダウンロードしました。
--2。ファイルマネージャーhttps://www.onworks.net/myfiles.php?username=XXXXXに必要なユーザー名を入力します。
-3。このアプリケーションをそのようなファイルマネージャにアップロードします。
-4。このWebサイトからOnWorksLinuxオンラインまたはWindowsオンラインエミュレーターまたはMACOSオンラインエミュレーターを起動します。
-5。起動したばかりのOnWorksLinux OSから、必要なユーザー名でファイルマネージャーhttps://www.onworks.net/myfiles.php?username=XXXXXにアクセスします。
-6。アプリケーションをダウンロードし、インストールして実行します。
スクリーンショットは
Ad
MANTI.pl / muda.pl
DESCRIPTION
--------注意の開始:リナミング
muda.plはv3.7でMANTI.plに名前が変更されました。プロジェクト開発は、のMANTIプロジェクトページで追跡できます。 sourceforge.net。 古いバージョンは、アーカイブの目的でここに残ります。
--------注意終了
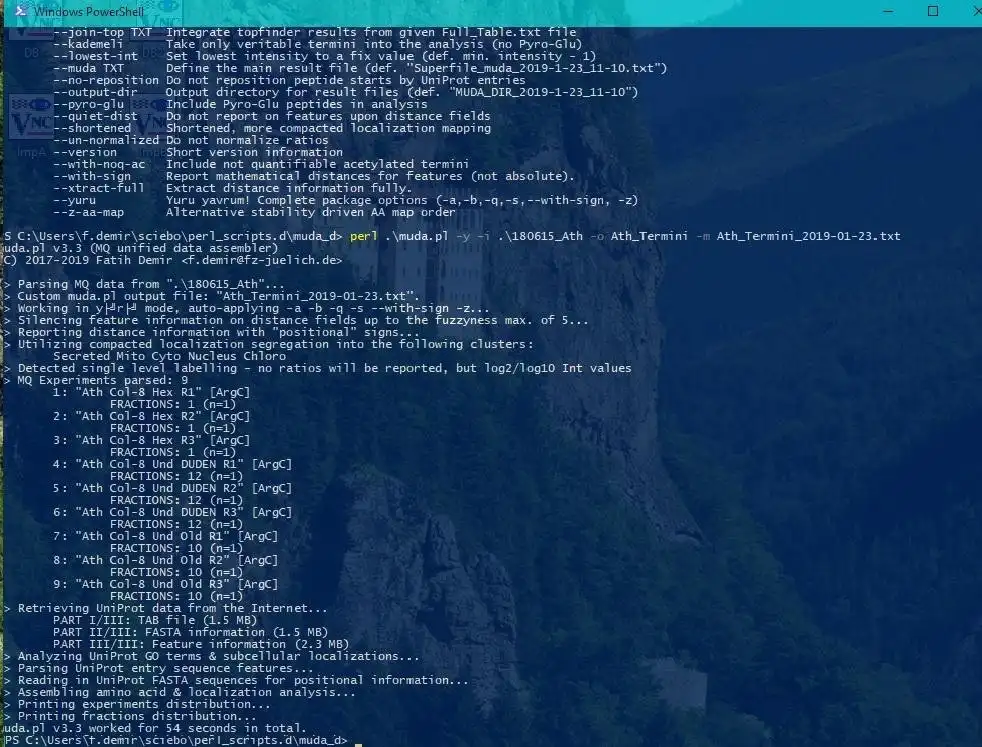
muda.plは、大きな依存関係のない評価スクリプト(Perlで記述)です。
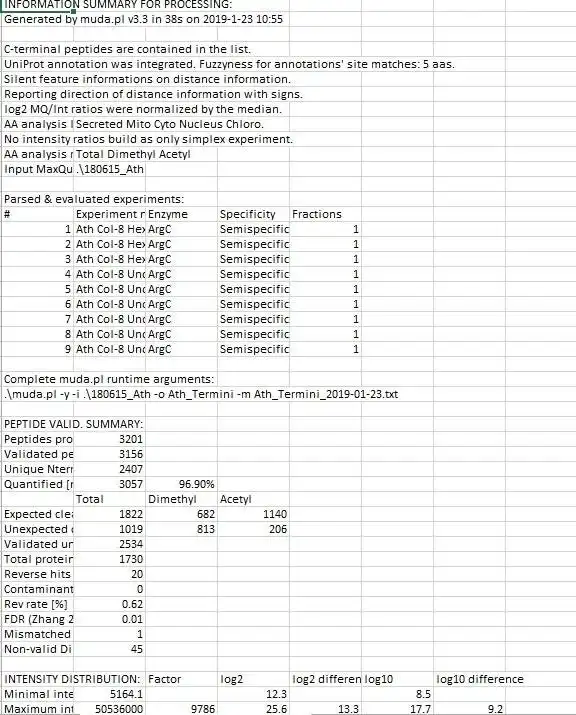
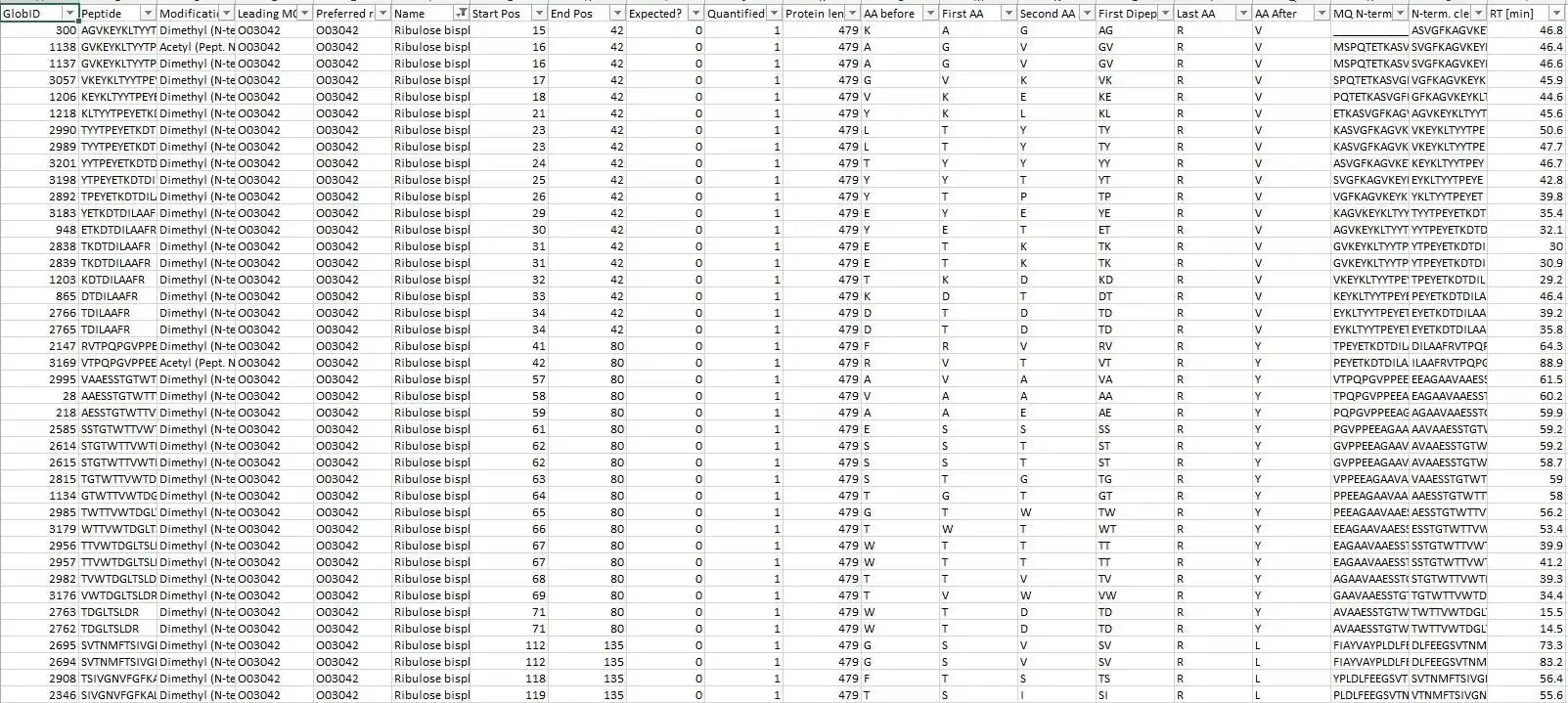
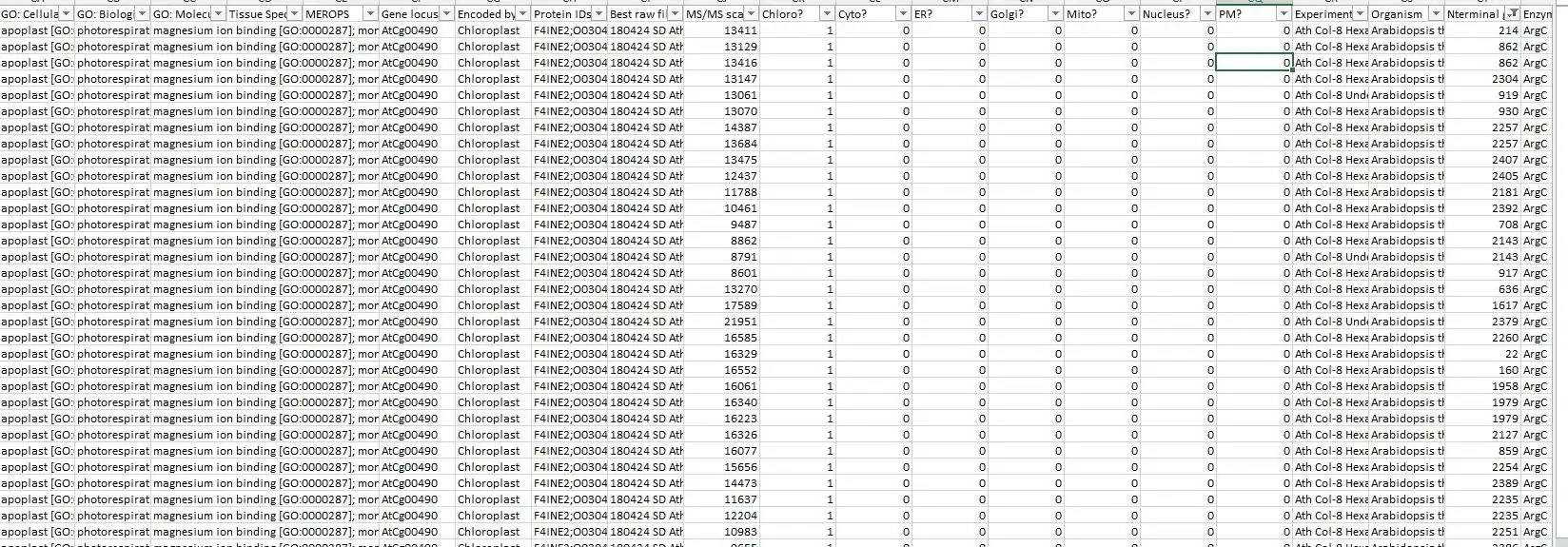
4つの異なるMaxQuant出力ファイルからの情報を、タンパク質のネオターミニ分析に明示的に適したマスターファイルに集約します。 データ集合の中心的なアンカーはmodificationSpecificPeptides.txtファイルです。追加のデータはMaxQuanttxtフォルダーの他のさまざまなソースファイルから推測されますが、データアセンブリの開始点はmodificationSpecificPeptides.txtファイルのみです。 通常のプロテオミクスの目的にも役立つ可能性がありますが、このスクリプトはタンパク質のネオ末端の同定と検証のために大幅に最適化されています。
スクリプトパラメータと評価戦略の詳細については、詳細なマニュアルPDFを参照してください。
特徴
- MaxQuantデータ評価
- タンパク質末端の同定と検証
- UniProtデータの注釈とアセンブリ
- 識別されたテルミニのアイソフォームへの再マッピング
- Limma、LOCALIZER、TopFINDerの統合
- 十分に文書化
Audience
科学/研究
ユーザーインターフェース
コマンドライン
プログラミング言語
パール
カテゴリー
これは、https://sourceforge.net/projects/muda/からも取得できるアプリケーションです。 無料のオペレーティングシステムのXNUMXつから最も簡単な方法でオンラインで実行するために、OnWorksでホストされています。