To jest aplikacja dla systemu Linux o nazwie TaxOnTree, której najnowszą wersję można pobrać jako TaxOnTree_v1.6.1.3_Linux_x86_64.tar.gz. Można go uruchomić online w bezpłatnym dostawcy hostingu OnWorks dla stacji roboczych.
Pobierz i uruchom online aplikację o nazwie TaxOnTree z OnWorks za darmo.
Postępuj zgodnie z tymi instrukcjami, aby uruchomić tę aplikację:
- 1. Pobrałem tę aplikację na swój komputer.
- 2. Wpisz w naszym menedżerze plików https://www.onworks.net/myfiles.php?username=XXXXX z wybraną nazwą użytkownika.
- 3. Prześlij tę aplikację w takim menedżerze plików.
- 4. Uruchom emulator online OnWorks Linux lub Windows online lub emulator online MACOS z tej witryny.
- 5. W systemie operacyjnym OnWorks Linux, który właśnie uruchomiłeś, przejdź do naszego menedżera plików https://www.onworks.net/myfiles.php?username=XXXXX z wybraną nazwą użytkownika.
- 6. Pobierz aplikację, zainstaluj ją i uruchom.
ZRZUTY EKRANU
Ad
Drzewo podatku
OPIS
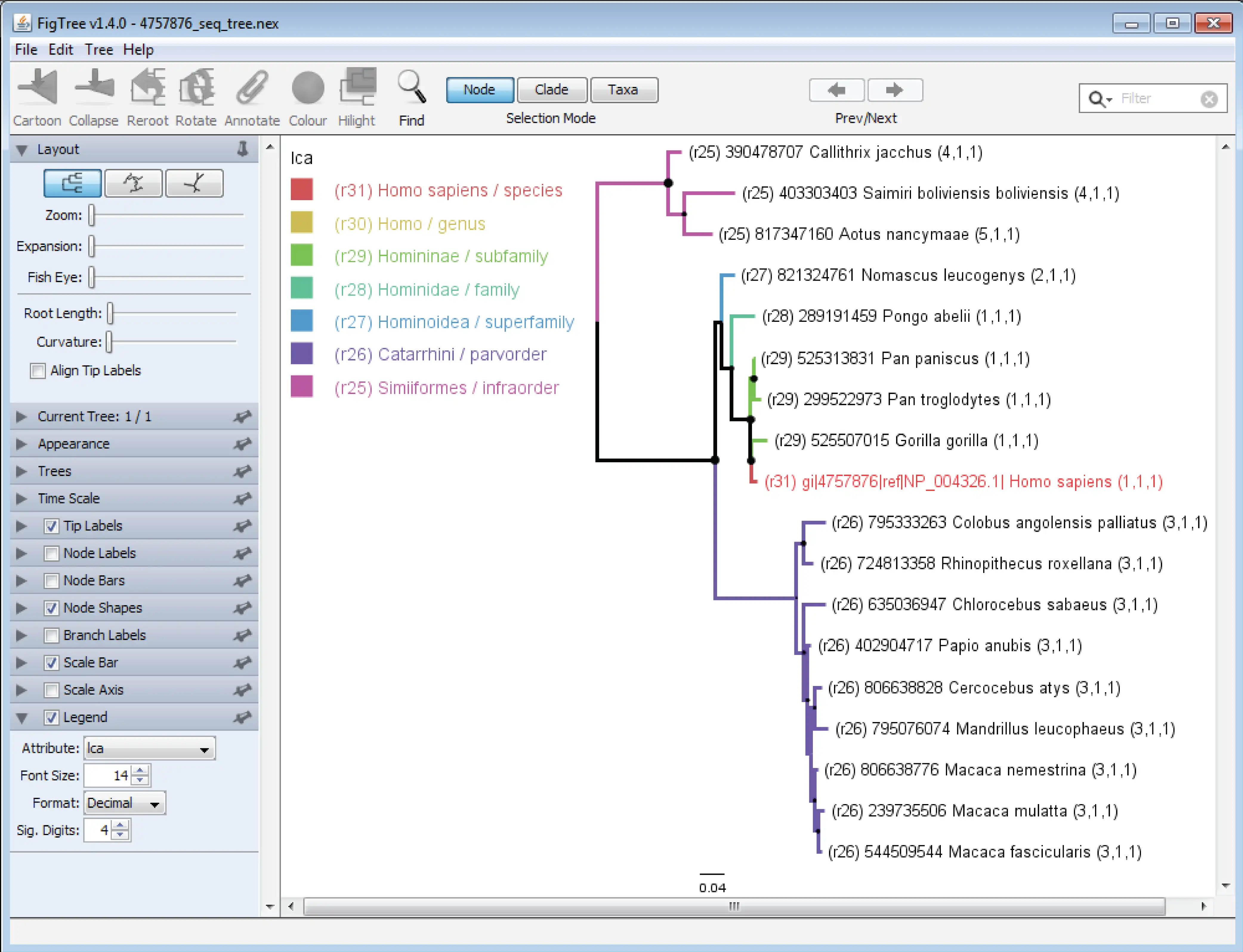
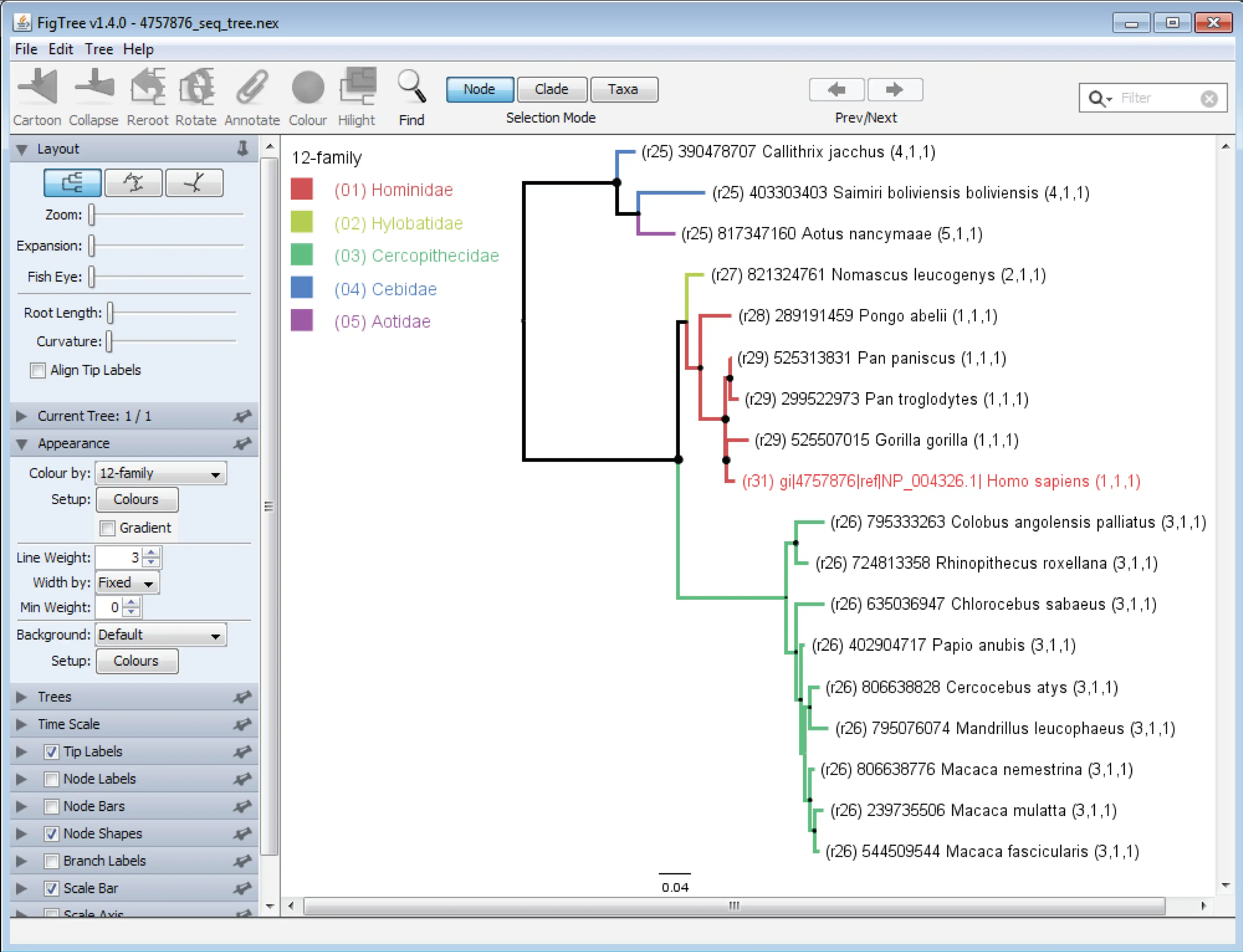
TaxOnTree to program filogenetyczny służący do łączenia informacji taksonomicznych w drzewie filogenetycznym.
Wynikiem jest plik drzewa w formacie NEX skonfigurowany do otwarcia w FigTree, który użytkownicy mogą szybko pokolorować według dowolnego taksonu lub pochodzenia wspólnego dla sekwencji za pomocą zapytania. Dane wejściowe mogą stanowić plik w formacie Fasta do wykorzystania w wyszukiwaniu BLAST lub lista sekwencji reprezentowanych przez ich identyfikatory (UniProtAC lub NCBI gi), jeśli klaster jest już dostępny. Ponadto plik newick utworzony za pomocą oprogramowania użytkownika i określonych ustawień może zostać wykorzystany do kontynuowania etykietowania taksonomicznego. TaxOnTree zmienia swojego użytkownika w eksperta w dziedzinie taksonomii. TaxOnTree koryguje również brak niektórych taksonów w taksonomii NCBI. Program wiersza poleceń jest łatwy w konfiguracji i można go połączyć z kilkoma narzędziami bioinformatycznymi, tworząc kolorowe drzewo w formacie PDF. TaxOnTree jest dostępny jako narzędzie internetowe w http://biodados.icb.ufmg.br/taxontree do mało wymagających prac.
Zakładka Charakterystyka
- Wprowadź zapytanie w formacie fasta (lub jego identyfikator) i wygeneruj drzewo filogenetyczne
- Baza danych RefSeq zawierająca wyłącznie kompletne białka i proteony UniProt Reference jest dystrybuowana wraz z TaxOnTree
- Wiele dopasowań można wykonać za pomocą MUSCLE, PRANK, Clustal Omega lub Kaliign
- Alternatywnie, jako dane wejściowe można wykorzystać klastry homologów, takie jak KO, jajkoNOG, OrthoMCL-DB, PANTHER itp.
- TaxOnTree może traktować wyrównanie za pomocą TrimAl i budować drzewo za pomocą FastTree
- Jednakże drzewo utworzone przez użytkownika w formacie Newick może być użyte jako dane wejściowe dla TaxOnTree
- TaxOnTree tworzy plik NEXUS, który można otworzyć za pomocą FigTree
- Aplikacja może uruchomić wiersz poleceń FigTree i wyeksportować analizowane drzewo do pliku PDF
- Do jednorazowego użytku dostępne jest narzędzie internetowe, którego kod jest również udostępniany do instalacji lokalnej
Publiczność
Nauka/Badania
Interfejs użytkownika
Wiersz poleceń
Język programowania
Perl
Środowisko bazy danych
Perl DBI/DBD, MySQL
Kategorie
Jest to aplikacja, którą można również pobrać ze strony https://sourceforge.net/projects/taxontree/. Został on hostowany w OnWorks, aby można go było uruchomić online w najprostszy sposób z jednego z naszych bezpłatnych systemów operacyjnych.