Este é o aplicativo Linux denominado gsasnp2 para rodar em Linux online, cuja versão mais recente pode ser baixada como gsasnp2-linux-cmd-ubuntu.zip. Ele pode ser executado online no provedor de hospedagem gratuita OnWorks para estações de trabalho.
Baixe e execute online este aplicativo chamado gsasnp2 para rodar em Linux online com OnWorks gratuitamente.
Siga estas instruções para executar este aplicativo:
- 1. Baixe este aplicativo em seu PC.
- 2. Entre em nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que você deseja.
- 3. Carregue este aplicativo em tal gerenciador de arquivos.
- 4. Inicie o emulador OnWorks Linux online ou Windows online ou emulador MACOS online a partir deste site.
- 5. No sistema operacional OnWorks Linux que você acabou de iniciar, acesse nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que deseja.
- 6. Baixe o aplicativo, instale-o e execute-o.
SCREENSHOTS
Ad
gsasnp2 para rodar em Linux online
DESCRIÇÃO
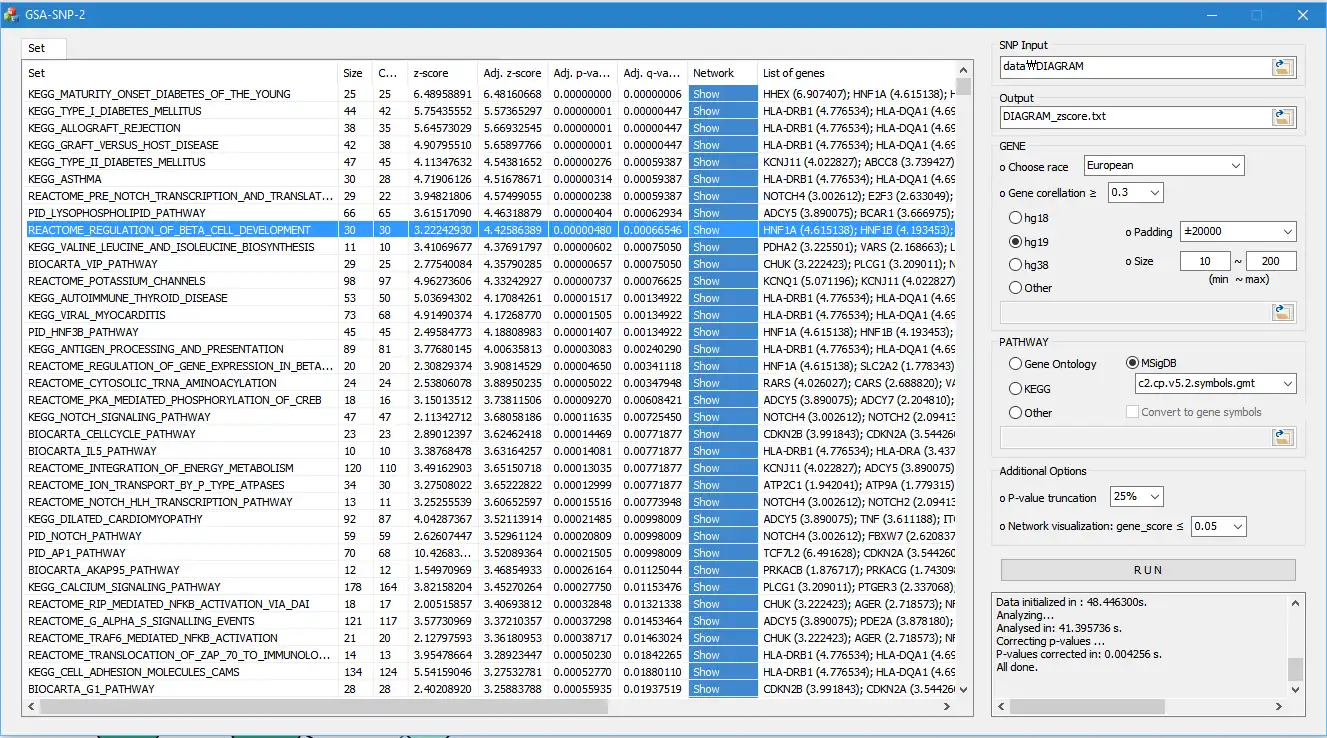
* GSA-SNP2 é um sucessor do GSA-SNP (Nam et al. 2010, NAR web server issue). GSA-SNP2 aceita dados resumidos de GWAS humanos (números rs, valores p) ou valores p genéticos e gera conjuntos de genes da via 'enriquecidos' com genes associados ao fenótipo fornecido. Ele também fornece redes de interação de proteínas locais e globais nas vias associadas.* Artigo: SYoon, HCTNguyen, YJYoo, JKim, BBaik, SKim, JKim, SKim, DNam, "Enriquecimento de via eficiente e análise de rede de dados resumidos de GWAS usando GSA-SNP2", Nucleic Acids Research, Vol. 46 (10), e60 (2018).
* ID PubMed: 29562348
* DOI: 10.1093 / nar / gky175
-> MOVA OU FAÇA UMA CÓPIA DA PASTA DE 'DADOS' PARA A SUA PASTA DE TESTE INTENSIVA (POR EXEMPLO, LINUX, MAC OU PASTA ESPECIFICADA DO WINDOWS) PARA PERMITIR QUE O PROGRAMA ENCONTRE OS DADOS PRÉ-DESIGNADOS.
* ATUALIZAÇÃO NOTA:
-> 7/2019/19.04: adicionar uma atualização para Ubuntu-XNUMX. Você precisará da biblioteca Boost instalada (sudo apt-get install libboost-all-dev)
-> Mar-7-2018: revisar os termos do cabeçalho no arquivo de saída
Recursos
- 1 / 'CONTROLE DE ERRO DECENTE TIPO I' obtido pelos dois processos a seguir: A) As pontuações dos genes são 'ajustadas' ao número de SNPs atribuídos a cada gene usando uma curva de tendência spline cúbica monótona. B) Genes adjacentes com altas correlações entre genes dentro de cada via foram removidos
- 2 / 'ALTA POTÊNCIA E COMPUTAÇÃO RÁPIDA' com base no modelo de conjunto aleatório
- 3 / 'SEM PARÂMETRO CRÍTICO LIVRE'
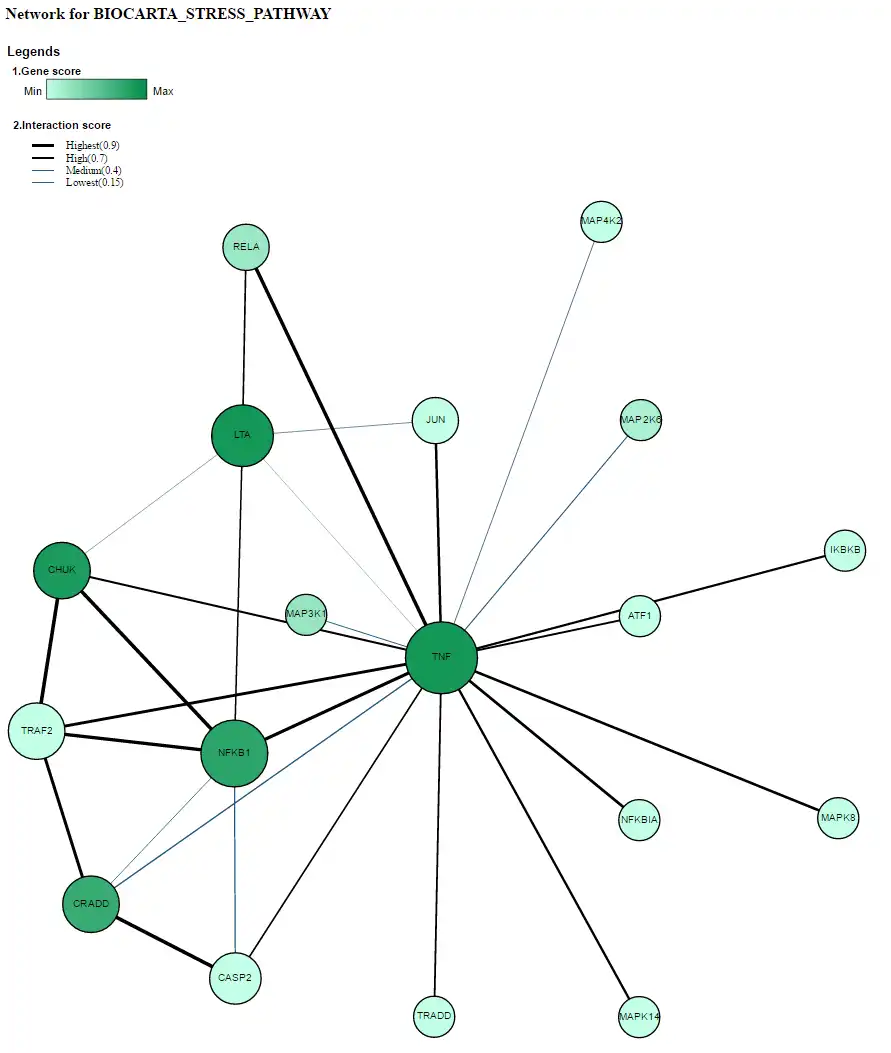
- 4 / 'REDES DE INTERAÇÃO DE PROTEÍNAS' entre os genes membros foram visualizados para as vias significativas. Esta função permite que o usuário priorize as sub-redes centrais dentro e entre os caminhos significativos. As redes STRING e HIPPIE são fornecidas atualmente
- 5 / 'FÁCIL DE USAR': Requer apenas dados de resumo GWAS (ou valores p do gene) e leva apenas um ou dois minutos para obter os resultados. Outras poderosas ferramentas de caminho autocontidas também requerem a entrada de correlação SNP e levam muito mais tempo. O usuário também pode fazer upload de seus próprios conjuntos de genes de vias e redes de interação de proteínas.
Interface com o usuário
Win32 (MS Windows), linha de comando
Linguagem de Programação
C ++, PHP, JavaScript
Este é um aplicativo que também pode ser obtido em https://sourceforge.net/projects/gsasnp2/. Ele foi hospedado no OnWorks para ser executado online da maneira mais fácil a partir de um de nossos Sistemas Operativos gratuitos.