Este é o aplicativo Linux chamado SPANDx cuja versão mais recente pode ser baixada como SPANDx_v3.2.tar.gz. Ele pode ser executado online no provedor de hospedagem gratuito OnWorks para estações de trabalho.
Baixe e execute online este aplicativo chamado SPANDx com OnWorks gratuitamente.
Siga estas instruções para executar este aplicativo:
- 1. Baixe este aplicativo em seu PC.
- 2. Entre em nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que você deseja.
- 3. Carregue este aplicativo em tal gerenciador de arquivos.
- 4. Inicie o emulador OnWorks Linux online ou Windows online ou emulador MACOS online a partir deste site.
- 5. No sistema operacional OnWorks Linux que você acabou de iniciar, acesse nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que deseja.
- 6. Baixe o aplicativo, instale-o e execute-o.
SCREENSHOTS
Ad
SPANDx
DESCRIÇÃO
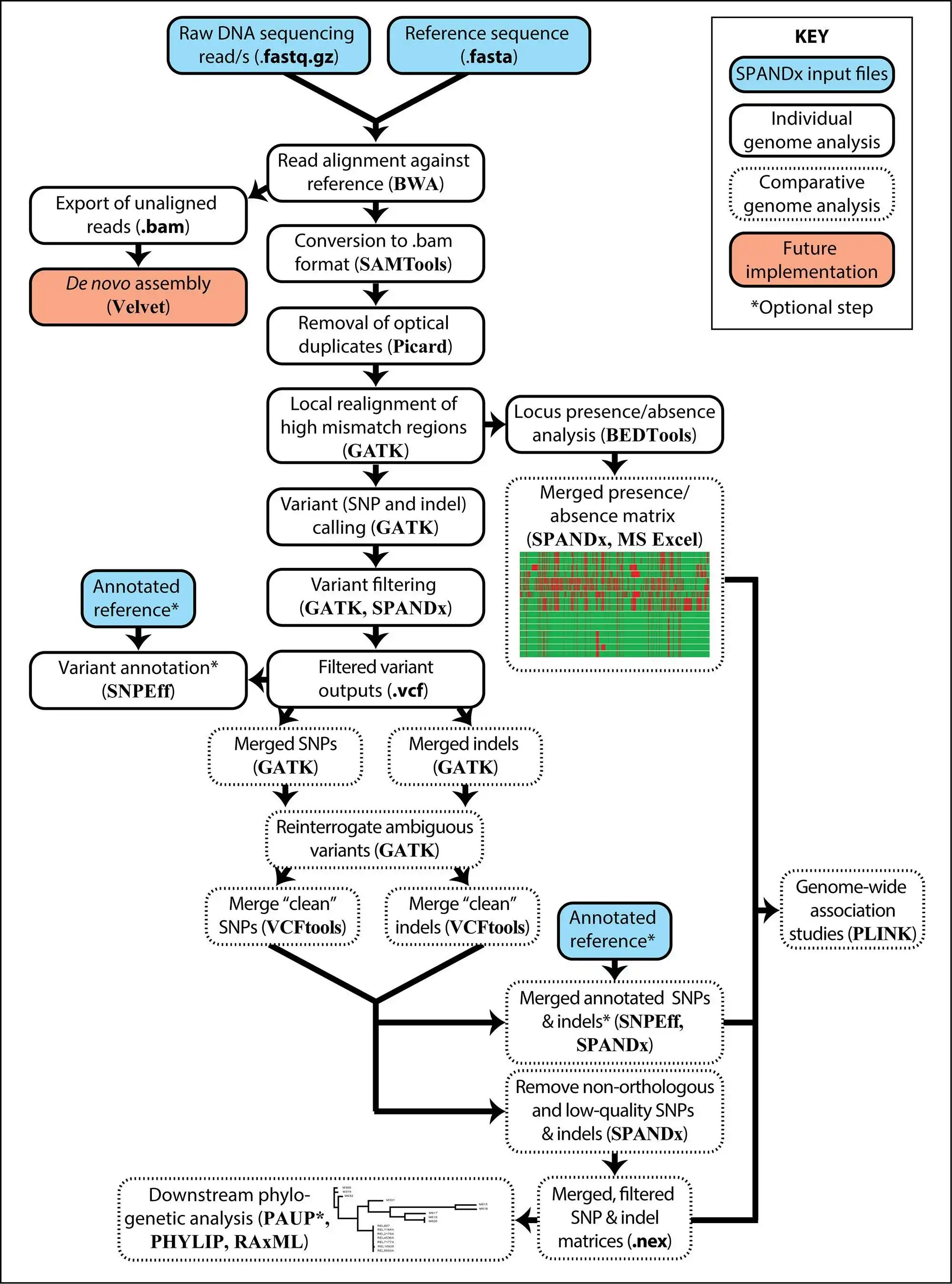
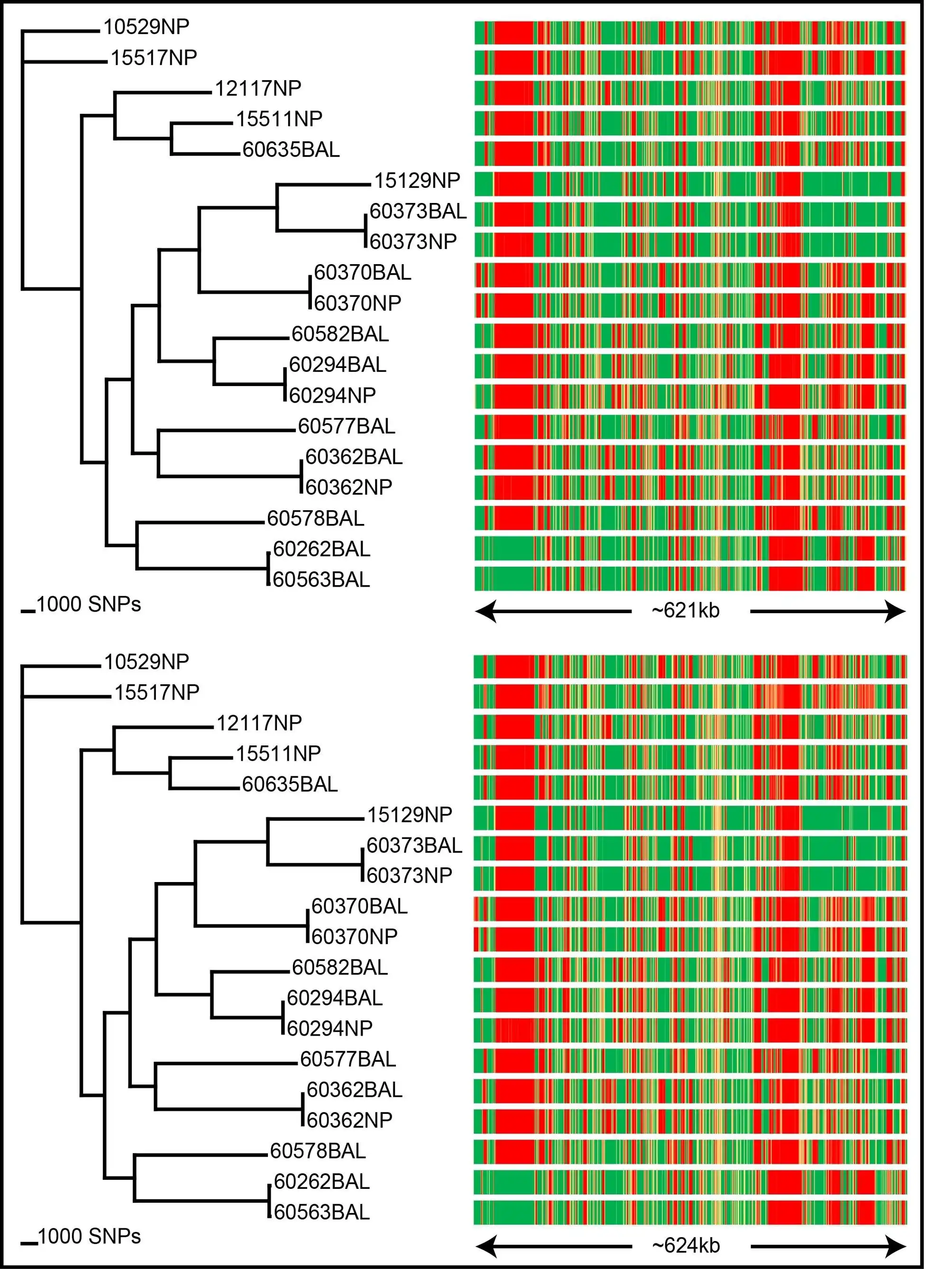
SPANDx é a sua ferramenta única para identificar SNP e variantes indel em genomas haplóides usando dados NGS.
O SPANDx realiza o alinhamento de leituras NGS brutas em relação ao genoma de referência ou pan-genoma escolhido, seguido por chamada e anotação de variantes precisas e determinação de presença / ausência de locus. SPANDx produz SNP e matrizes indel para análises filogenéticas downstream. SNPs e indels anotados em todo o genoma também podem ser identificados, se especificados, e são produzidos em formato legível por humanos. Uma matriz de presença / ausência também é gerada para permitir que você identifique o conteúdo do genoma central / acessório em todos os seus genomas.
Os resultados gerados pelo SPANDx podem ser importados para o PLINK para análises de estudo de associação do genoma microbiano (mGWAS).
SPANDx pode utilizar PBS, SGE ou SLURM para gerenciamento de recursos. O SPANDx também pode ser executado diretamente na linha de comando se nenhum gerenciador de recursos estiver disponível.
Para obter a versão mais atualizada do SPANDx, verifique-nos no GitHub: https://github.com/dsarov/SPANDx
Funcionalidades
- 30Nov16: SPANDx 3.2 agora usa BWA-mem para alinhamentos mais rápidos e precisos
- 31 de maio de 16: SPANDx 3.1.1 agora funciona em sistemas TORQUE / PBS pré-v2.3.1.
- 14Fev16: SPANDx 3.1 O sinalizador -z agora pode ser definido para incluir SNPs tri e tetra-alélicos nas saídas .nex.
- 26Dec15: SPANDx 3.0 tem muitas atualizações, incluindo compatibilidade melhorada com samtools, melhor integração PLINK para GWAS e pode chamar até 9 indels e todos os quatro SNPs em um único locus!
- 05 de agosto: SPANDx v15 agora funciona em SGE, PBS, SLURM e sistemas sem um gerenciador de recursos.
Público
Ciência / Pesquisa
Interface com o usuário
Linha de comando
Linguagem de Programação
Shell Unix
Categorias
Este é um aplicativo que também pode ser obtido em https://sourceforge.net/projects/spandx/. Ele foi hospedado no OnWorks para ser executado online da maneira mais fácil a partir de um de nossos Sistemas Operativos gratuitos.