Este é o aplicativo do Windows chamado GenomeRunner, cuja versão mais recente pode ser baixada como GenomeRunner-v4.0-Setup.zip. Ele pode ser executado online no provedor de hospedagem gratuito OnWorks para estações de trabalho.
Baixe e execute online este aplicativo chamado GenomeRunner com OnWorks gratuitamente.
Siga estas instruções para executar este aplicativo:
- 1. Baixe este aplicativo em seu PC.
- 2. Entre em nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que você deseja.
- 3. Carregue este aplicativo em tal gerenciador de arquivos.
- 4. Inicie qualquer emulador on-line OS OnWorks a partir deste site, mas um emulador on-line melhor do Windows.
- 5. No sistema operacional OnWorks Windows que você acabou de iniciar, acesse nosso gerenciador de arquivos https://www.onworks.net/myfiles.php?username=XXXXX com o nome de usuário que deseja.
- 6. Baixe o aplicativo e instale-o.
- 7. Baixe o Wine de seus repositórios de software de distribuição Linux. Depois de instalado, você pode clicar duas vezes no aplicativo para executá-lo com o Wine. Você também pode experimentar o PlayOnLinux, uma interface sofisticada do Wine que o ajudará a instalar programas e jogos populares do Windows.
Wine é uma forma de executar software Windows no Linux, mas sem a necessidade de Windows. Wine é uma camada de compatibilidade do Windows de código aberto que pode executar programas do Windows diretamente em qualquer desktop Linux. Essencialmente, o Wine está tentando reimplementar o suficiente do Windows do zero para que possa executar todos os aplicativos do Windows sem realmente precisar do Windows.
SCREENSHOTS
Ad
GenomeRunner
DESCRIÇÃO
Nota: Esta versão requer arquivos de banco de dados SQLite adicionais. Entre em contato com os desenvolvedores para obtê-los.
Use http://www.integrativegenomics.org/ para obter os dados e análises mais recentes.
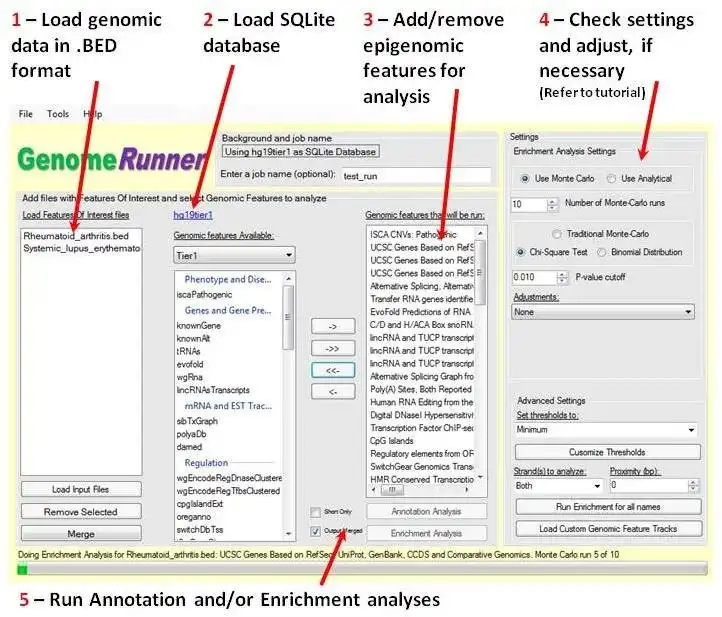
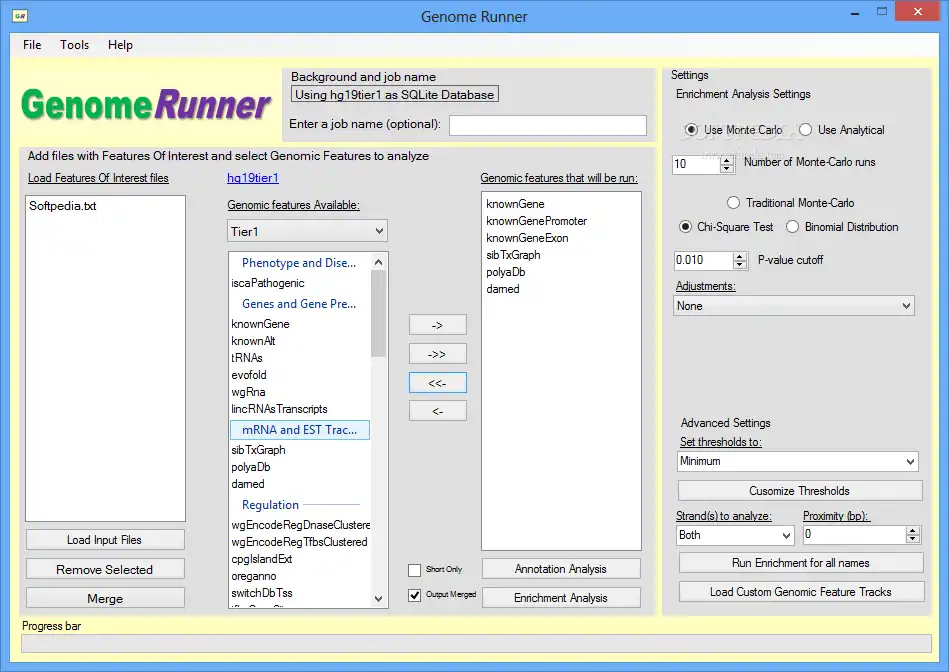
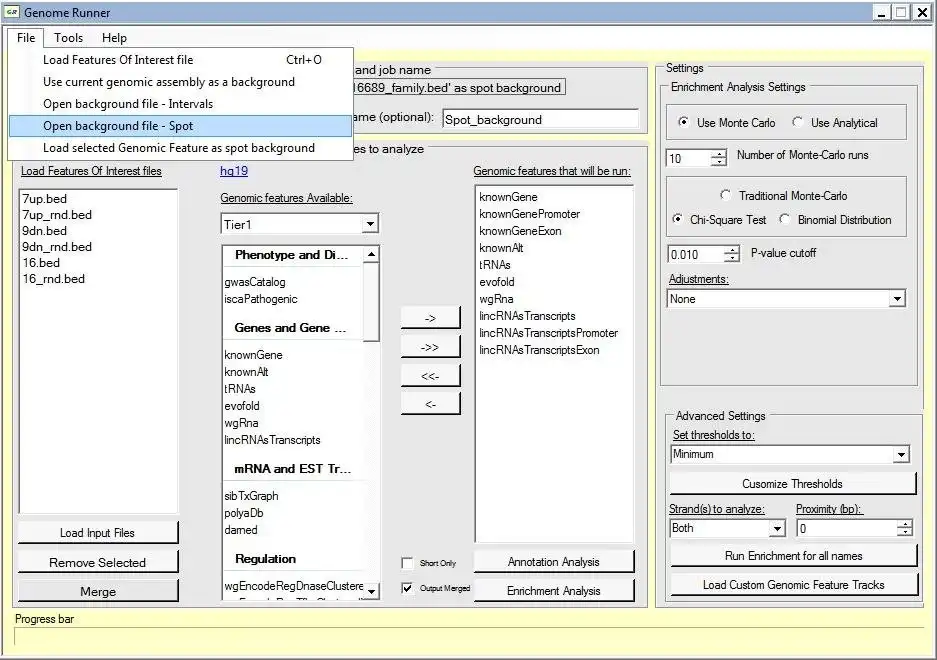
GenomeRunner é uma ferramenta para automatizar a exploração do genoma. Ele executa análises de anotação e enriquecimento de regiões genômicas fornecidas pelo usuário (SNPs, sítios de ligação ChIP-seq etc.) contra mais de 6,000 (genoma humano) recursos epigenômicos disponíveis no navegador de genoma UCSC.
Entrada - quaisquer dados de dados de todo o genoma no formato .bed (arquivo de texto delimitado por tabulação com chrom, chromStart, chromEnd).
Saída de análise de anotação - anotação detalhada de cada região genômica nos dados de entrada. Usado para priorizar regiões genômicas individuais pelo número total de características epigenômicas com as quais elas se localizam.
Saída da análise de enriquecimento - valores p de colocalizações estatisticamente significativas de dados de entrada de todo o genoma com recursos de anotação de genoma selecionados para a análise. Usado para priorizar recursos epigenômicos associados aos dados do usuário.
Recursos
- Análise de regiões genômicas (ChIP-seq, RNA-seq, metilação de DNA, SNPs/CNVs etc.)
- CODIFICAR dados
- Análises de anotação e enriquecimento
- Formato de arquivo BED (chrom, chromStart, chromEnd)
- Visão geral do vídeo (http://youtu.be/v9p9FClrqXU)
Público
Ciência / Pesquisa, Usuários Finais / Desktop
Interface com o usuário
O projeto é um sistema de interface do usuário (IU)
Linguagem de Programação
Visual Basic .NET
Ambiente de Banco de Dados
Baseado em SQL
Categorias
Este é um aplicativo que também pode ser obtido em https://sourceforge.net/projects/genomerunner/. Ele foi hospedado no OnWorks para ser executado on-line da maneira mais fácil a partir de um de nossos sistemas operacionais gratuitos.