Это приложение для Linux с именем gsasnp2, последнюю версию которого можно загрузить как gsasnp2-linux-cmd-ubuntu.zip. Его можно запустить онлайн на бесплатном хостинг-провайдере OnWorks для рабочих станций.
Загрузите и запустите онлайн это приложение под названием gsasnp2 с OnWorks бесплатно.
Следуйте этим инструкциям, чтобы запустить это приложение:
- 1. Загрузил это приложение на свой компьютер.
- 2. Введите в нашем файловом менеджере https://www.onworks.net/myfiles.php?username=XXXXX с желаемым именем пользователя.
- 3. Загрузите это приложение в такой файловый менеджер.
- 4. Запустите онлайн-эмулятор OnWorks Linux или Windows или онлайн-эмулятор MACOS с этого веб-сайта.
- 5. В только что запущенной ОС OnWorks Linux перейдите в наш файловый менеджер https://www.onworks.net/myfiles.php?username=XXXXX с желаемым именем пользователя.
- 6. Скачайте приложение, установите его и запустите.
СКРИНШОТЫ
Ad
gsasnp2
ОПИСАНИЕ
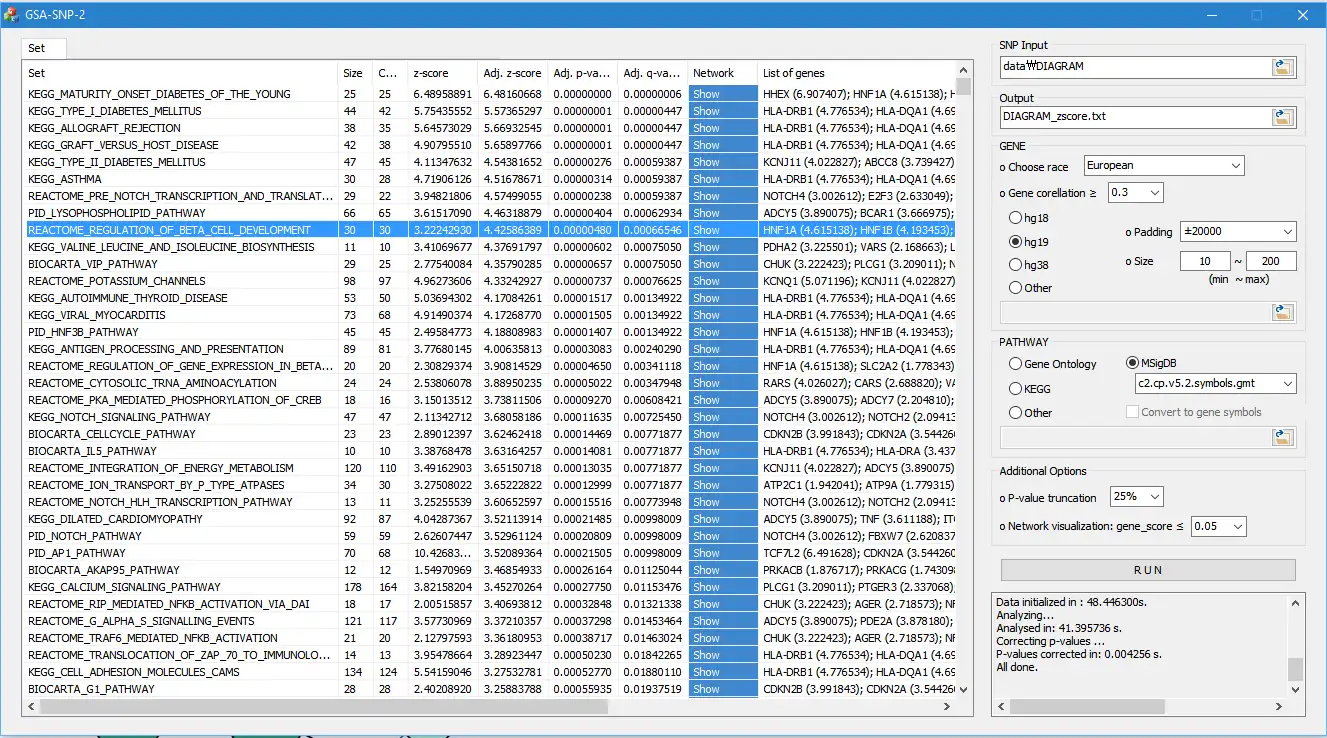
* GSA-SNP2 является преемником GSA-SNP (Nam et al. 2010, выпуск веб-сервера NAR). GSA-SNP2 принимает сводные данные GWAS человека (числа rs, p-значения) или генные значения p и выводит наборы генов, «обогащенные» генами, связанными с данным фенотипом. Он также обеспечивает как локальные, так и глобальные сети взаимодействия белков в связанных путях.
* Статья: SYoon, HCTNguyen, YJYoo, JKim, BBaik, SKim, JKim, SKim, DNam, «Эффективное обогащение путей и сетевой анализ сводных данных GWAS с использованием GSA-SNP2», Nucleic Acids Research, Vol. 46 (10), e60 (2018).
* Идентификатор PubMed: 29562348
* DOI: 10.1093 / nar / gky175
-> ПОЖАЛУЙСТА, ПЕРЕМЕСТИТЕ ИЛИ СДЕЛАЙТЕ КОПИЮ ПАПКИ «ДАННЫЕ» В ВАШУ ИНТЕНСИВНУЮ ТЕСТОВУЮ ПАПКУ (ПАПКА, УКАЗАННАЯ IE LINUX, MAC ИЛИ WINDOWS), ЧТОБЫ РАЗРЕШИТЬ ПРОГРАММУ НАЙТИ ПРЕДНАЗНАЧЕННЫЕ ДАННЫЕ.
* ОБНОВЛЕНИЕ ПРИМЕЧАНИЕ:
-> 1 сентября 2020 г.: добавить обновление для Ubuntu-20.04. Вам понадобится установленная библиотека Boost (sudo apt-get install libboost-all-dev)
-> Март-7-2018: изменить условия заголовка в выходном файле
Особенности
- 1 / «ДОСТОЙНОЕ УПРАВЛЕНИЕ ОШИБКАМИ ТИПА I» достигается с помощью следующих двух процессов: A) Показатели генов «корректируются» в соответствии с количеством SNP, назначенных каждому гену, с использованием монотонной кубической сплайновой кривой тренда. Б) Смежные гены с высокими межгенными корреляциями внутри каждого пути были удалены.
- 2 / «ВЫСОКАЯ МОЩНОСТЬ И БЫСТРЫЕ ВЫЧИСЛЕНИЯ» на основе модели случайного набора
- 3 / «ОТСУТСТВИЕ КРИТИЧЕСКИХ СВОБОДНЫХ ПАРАМЕТРОВ»
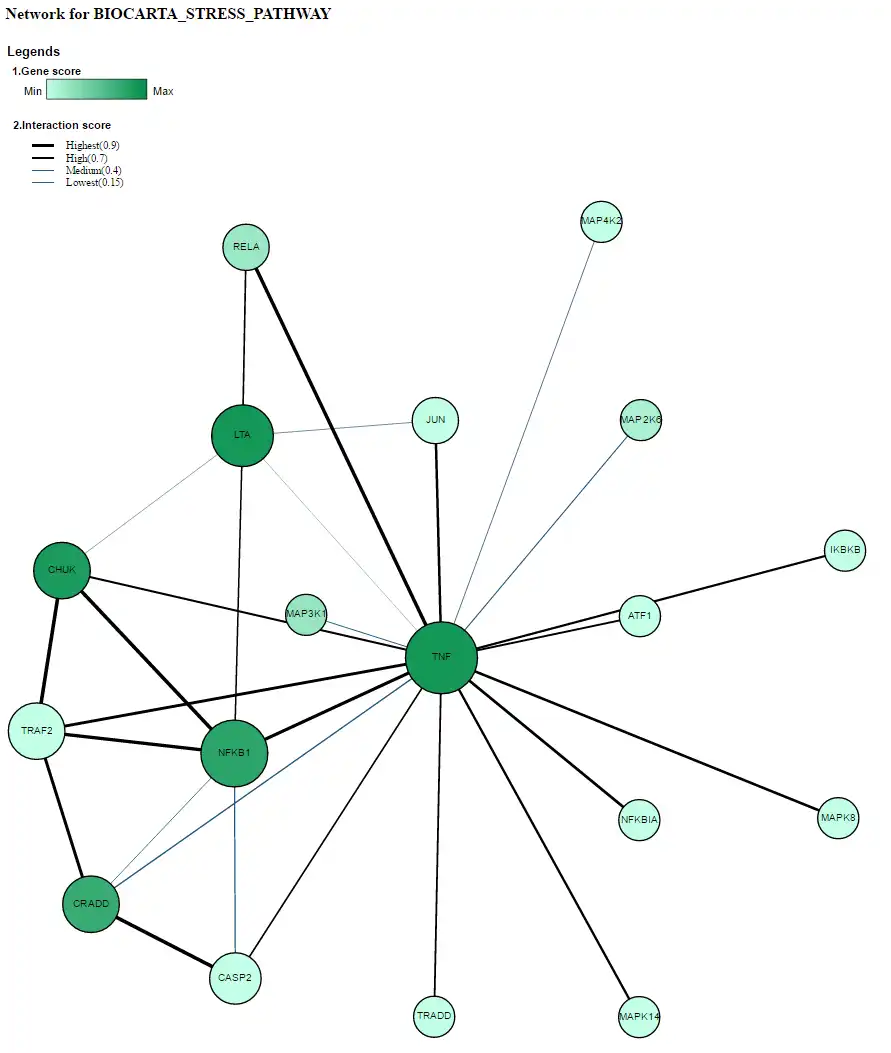
- 4 / «СЕТИ ВЗАИМОДЕЙСТВИЙ С БЕЛКАМИ» среди генов-членов были визуализированы для важных путей. Эта функция позволяет пользователю определять приоритеты основных подсетей внутри и между важными маршрутами. В настоящее время доступны сети STRING и HIPPIE.
- 5 / «ПРОСТОТА ИСПОЛЬЗОВАНИЯ»: для этого требуются только сводные данные GWAS (или p-значения генов), и для получения результатов требуется всего одна или две минуты. Другие мощные автономные инструменты пути также требуют ввода корреляции SNP и занимают гораздо больше времени. Пользователь также может загружать свои собственные наборы генов путей и сети взаимодействия белков.
Интерфейс пользователя
Win32 (MS Windows), командная строка
Язык программирования
C ++, PHP, JavaScript
Категории
Это приложение также можно загрузить с https://sourceforge.net/projects/gsasnp2/. Он размещен в OnWorks, чтобы его можно было легко запускать в сети с помощью одной из наших бесплатных операционных систем.