Это приложение для Linux с именем SPANDx, последний выпуск которого можно загрузить как SPAANDx_v3.2.tar.gz. Его можно запустить онлайн на бесплатном хостинг-провайдере OnWorks для рабочих станций.
Загрузите и запустите онлайн это приложение под названием SPANDx с OnWorks бесплатно.
Следуйте этим инструкциям, чтобы запустить это приложение:
- 1. Загрузил это приложение на свой компьютер.
- 2. Введите в нашем файловом менеджере https://www.onworks.net/myfiles.php?username=XXXXX с желаемым именем пользователя.
- 3. Загрузите это приложение в такой файловый менеджер.
- 4. Запустите онлайн-эмулятор OnWorks Linux или Windows или онлайн-эмулятор MACOS с этого веб-сайта.
- 5. В только что запущенной ОС OnWorks Linux перейдите в наш файловый менеджер https://www.onworks.net/myfiles.php?username=XXXXX с желаемым именем пользователя.
- 6. Скачайте приложение, установите его и запустите.
СКРИНШОТЫ
Ad
СПАНДкс
ОПИСАНИЕ
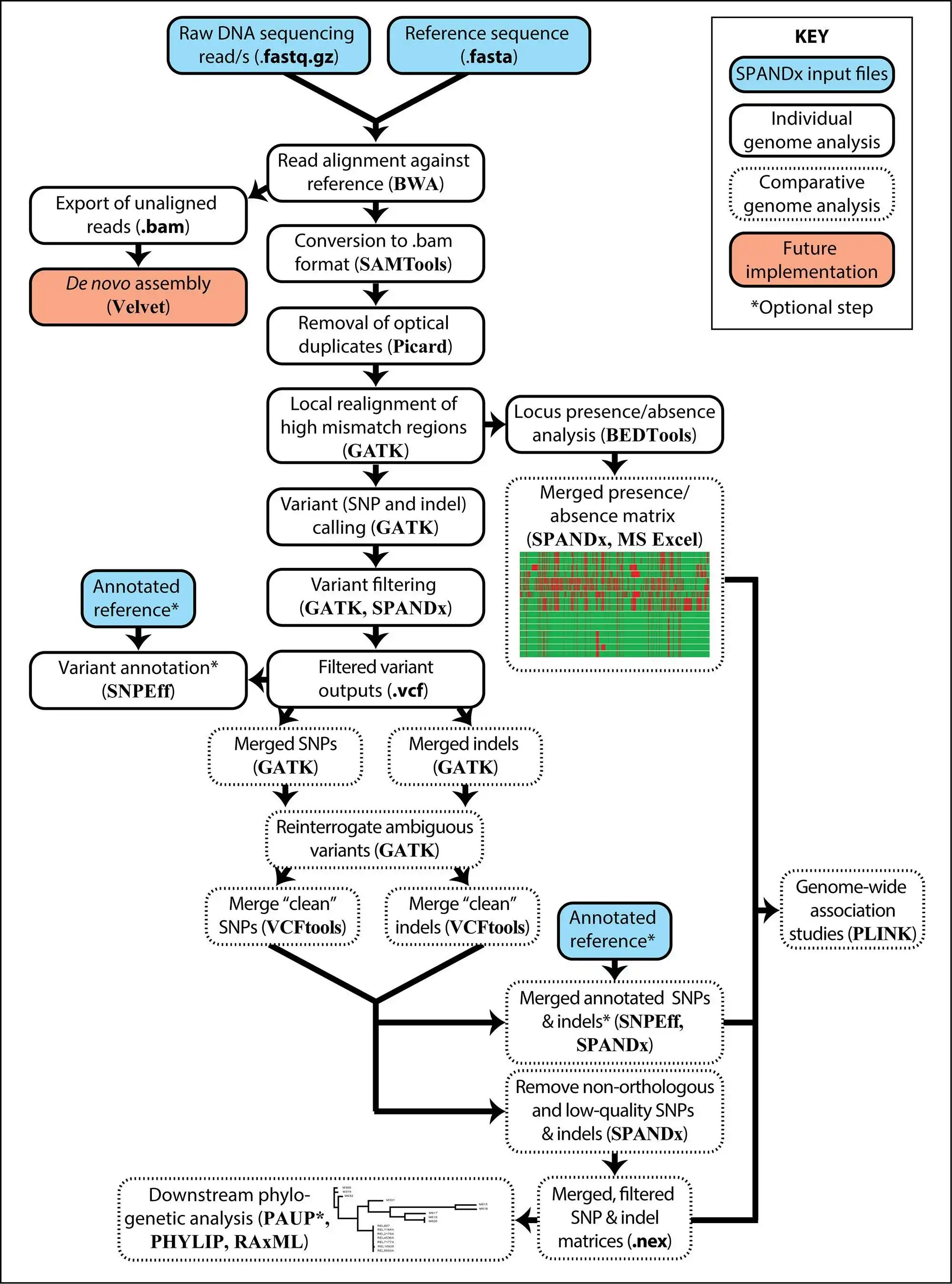
SPANDx - ваш универсальный инструмент для идентификации вариантов SNP и indel в гаплоидных геномах с использованием данных NGS.
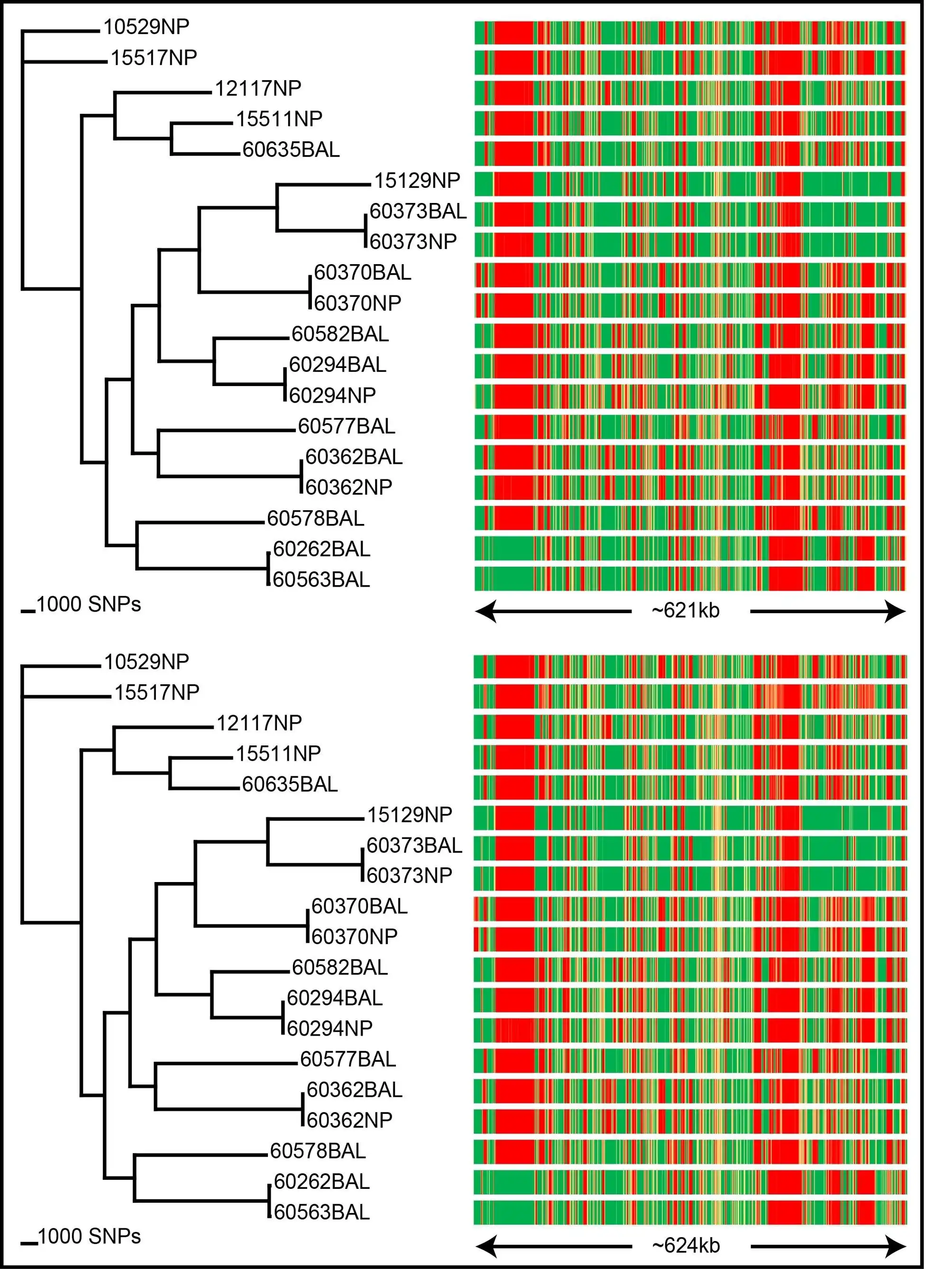
SPANDx выполняет выравнивание необработанных считываний NGS с выбранным вами эталонным геномом или пангеномом с последующим точным вызовом и аннотацией вариантов, а также определением наличия / отсутствия локуса. SPANDx производит матрицы SNP и indel для последующего филогенетического анализа. Аннотированные, общегеномные SNP и вставки также могут быть идентифицированы, если указаны, и выводятся в удобочитаемом формате. Также создается матрица присутствия / отсутствия, позволяющая идентифицировать содержание основного / вспомогательного генома во всех ваших геномах.
Результаты, генерируемые SPANDx, могут быть импортированы в PLINK для анализа микробных ассоциаций генома (mGWAS).
SPANDx может использовать PBS, SGE или SLURM для управления ресурсами. SPANDx также может запускаться непосредственно из командной строки, если диспетчер ресурсов недоступен.
Самую последнюю версию SPANDx вы можете найти на GitHub: https://github.com/dsarov/SPANDx
Особенности
- 30Nov16: SPANDx 3.2 теперь использует BWA-mem для более быстрого и точного выравнивания
- 31 мая, 16: SPANDx 3.1.1 теперь работает в системах TORQUE / PBS до версии 2.3.1.
- 14Feb16: SPANDx 3.1 Теперь можно установить флаг -z для включения три- и тетрааллельных SNP в выходные данные .nex.
- 26 декабря 15 года: SPANDx 3.0 имеет множество обновлений, включая улучшенную совместимость с Samtools, лучшую интеграцию PLINK для GWAS, и может вызывать до 9 индексов и все четыре SNP на одном локусе!
- 05 августа 15: SPANDx v2.7 теперь работает на SGE, PBS, SLURM и системах без диспетчера ресурсов.
Аудитория
Наука / Исследования
Интерфейс пользователя
Командная строка
Язык программирования
Оболочка Unix
Категории
Это приложение также можно загрузить с https://sourceforge.net/projects/spandx/. Он размещен в OnWorks, чтобы его можно было легко запускать в Интернете с помощью одной из наших бесплатных операционных систем.