这是一个名为 gsasnp2 的 Windows 应用程序,可在 Windows 上在线运行,其最新版本可以下载为 gsasnp2-linux-cmd-ubuntu.zip。 它可以在工作站的免费托管服务提供商 OnWorks 中在线运行。
下载并在线运行这个名为 gsasnp2 的应用程序,可通过 OnWorks 免费在线在 Windows 上运行。
请按照以下说明运行此应用程序:
- 1. 在您的 PC 中下载此应用程序。
- 2. 在我们的文件管理器 https://www.onworks.net/myfiles.php?username=XXXXX 中输入您想要的用户名。
- 3. 在这样的文件管理器中上传这个应用程序。
- 4. 从本网站启动任何 OS OnWorks 在线模拟器,但更好的 Windows 在线模拟器。
- 5. 从您刚刚启动的 OnWorks Windows 操作系统,使用您想要的用户名转到我们的文件管理器 https://www.onworks.net/myfiles.php?username=XXXXX。
- 6. 下载应用程序并安装。
- 7. 从您的 Linux 发行版软件存储库下载 Wine。 安装后,您可以双击该应用程序以使用 Wine 运行它们。 您还可以尝试 PlayOnLinux,这是 Wine 上的一个花哨界面,可帮助您安装流行的 Windows 程序和游戏。
Wine 是一种在 Linux 上运行 Windows 软件的方法,但不需要 Windows。 Wine 是一个开源的 Windows 兼容层,可以直接在任何 Linux 桌面上运行 Windows 程序。 本质上,Wine 试图从头开始重新实现足够多的 Windows,以便它可以运行所有这些 Windows 应用程序,而实际上不需要 Windows。
SCREENSHOTS
Ad
gsasnp2 在 Windows 上在线运行于 Linux 在线
商品描述
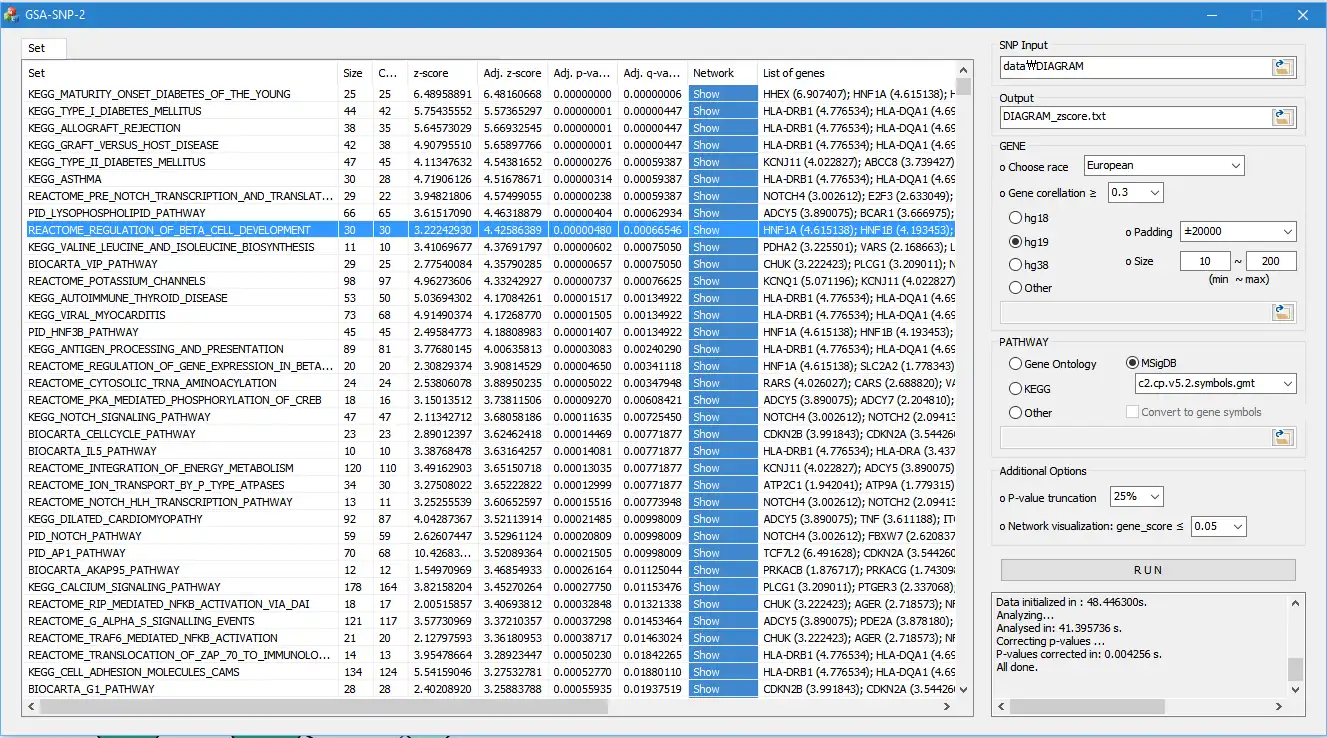
* GSA-SNP2 是 GSA-SNP 的继承者(Nam et al. 2010,NAR web server issue)。 GSA-SNP2 接受人类 GWAS 汇总数据(rs 数、p 值)或基因方面的 p 值,并输出“富含”与给定表型相关的基因的通路基因集。 它还提供相关途径中的局部和全局蛋白质相互作用网络。* 文章:SYoon、HCTNguyen、YJYoo、JKim、BBaik、SKim、JKim、SKim、DNam,“使用 GSA-SNP2 对 GWAS 汇总数据进行有效的通路富集和网络分析”,核酸研究,卷。 46(10),e60(2018)。
* PubMed ID:29562348
* DOI:10.1093/nar/gky175
-> 请将“数据”文件夹移动或复制到您的密集测试文件夹(即 LINUX、MAC 或 WINDOWS 指定文件夹)中,以便程序找到预先设计的数据。
* 更新注意:
-> 7 年 2019 月 19.04 日:为 Ubuntu-XNUMX 添加更新。 您将需要安装 Boost 库(sudo apt-get install libboost-all-dev)
-> Mar-7-2018:修改输出文件中的标题术语
产品优势
- 1/ 通过以下两个过程实现“DECENT TYPE I 错误控制”: A) 使用单调三次样条趋势曲线将基因分数“调整”到分配给每个基因的 SNP 数量。 B) 去除每个通路中具有高基因间相关性的相邻基因
- 2/ 基于随机集模型的“高功率和快速计算”
- 3/ '无关键自由参数'
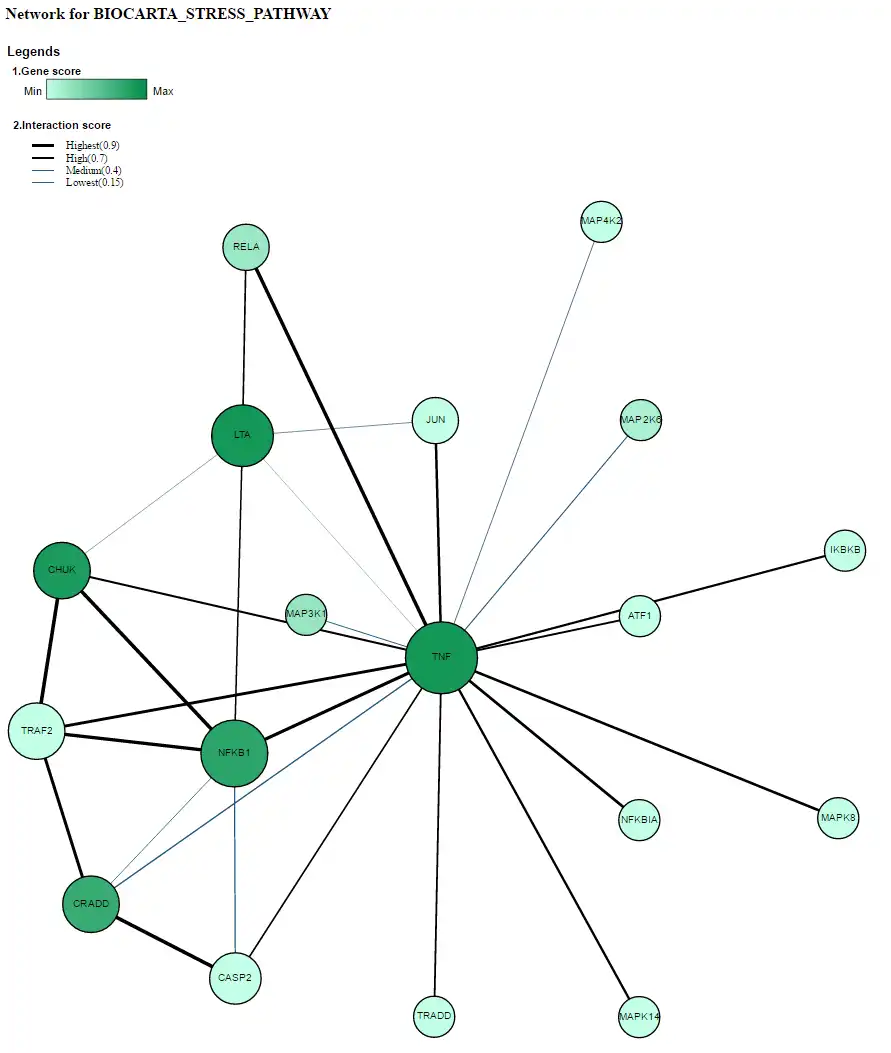
- 4/ 成员基因之间的“蛋白质相互作用网络”对于重要途径进行了可视化。 此功能使用户能够对重要路径内和重要路径之间的核心子网进行优先级排序。 目前提供STRING和HIPPIE网络
- 5/“易于使用”:它只需要 GWAS 汇总数据(或基因 p 值),只需一两分钟即可获得结果。 其他强大的自包含通路工具也需要 SNP 相关输入,并且需要更长的时间。 用户还可以上传他们自己的通路基因集和蛋白质相互作用网络。
用户界面
Win32 (MS Windows),命令行
程式语言
C++、PHP、JavaScript
这是一个也可以从 https://sourceforge.net/projects/gsasnp2/ 获取的应用程序。 它已托管在 OnWorks 中,以便从我们的免费操作系统之一以最简单的方式在线运行。