Dies ist die Windows-App namens Visualization of Protein-Ligand Graphs, die unter Windows online über Linux online ausgeführt werden kann. Die neueste Version kann als vplg_2016_07_18.zip heruntergeladen werden. Es kann online beim kostenlosen Hosting-Anbieter OnWorks für Workstations ausgeführt werden.
Laden Sie diese App mit dem Namen Visualization of Protein-Ligand Graphs herunter und führen Sie sie online aus, um sie unter Windows online über Linux online mit OnWorks kostenlos auszuführen.
Befolgen Sie diese Anweisungen, um diese App auszuführen:
- 1. Diese Anwendung auf Ihren PC heruntergeladen.
- 2. Geben Sie in unserem Dateimanager https://www.onworks.net/myfiles.php?username=XXXXX den gewünschten Benutzernamen ein.
- 3. Laden Sie diese Anwendung in einem solchen Dateimanager hoch.
- 4. Starten Sie einen beliebigen OS OnWorks-Online-Emulator von dieser Website, aber einen besseren Windows-Online-Emulator.
- 5. Rufen Sie vom gerade gestarteten OnWorks Windows-Betriebssystem unseren Dateimanager https://www.onworks.net/myfiles.php?username=XXXXX mit dem gewünschten Benutzernamen auf.
- 6. Laden Sie die Anwendung herunter und installieren Sie sie.
- 7. Laden Sie Wine aus den Software-Repositorys Ihrer Linux-Distributionen herunter. Nach der Installation können Sie dann auf die App doppelklicken, um sie mit Wine auszuführen. Sie können auch PlayOnLinux ausprobieren, eine schicke Schnittstelle über Wine, die Ihnen bei der Installation beliebter Windows-Programme und -Spiele hilft.
Wine ist eine Möglichkeit, Windows-Software unter Linux auszuführen, jedoch ohne Windows. Wine ist eine Open-Source-Windows-Kompatibilitätsschicht, die Windows-Programme direkt auf jedem Linux-Desktop ausführen kann. Im Wesentlichen versucht Wine, genügend Windows von Grund auf neu zu implementieren, damit alle diese Windows-Anwendungen ausgeführt werden können, ohne dass Windows tatsächlich benötigt wird.
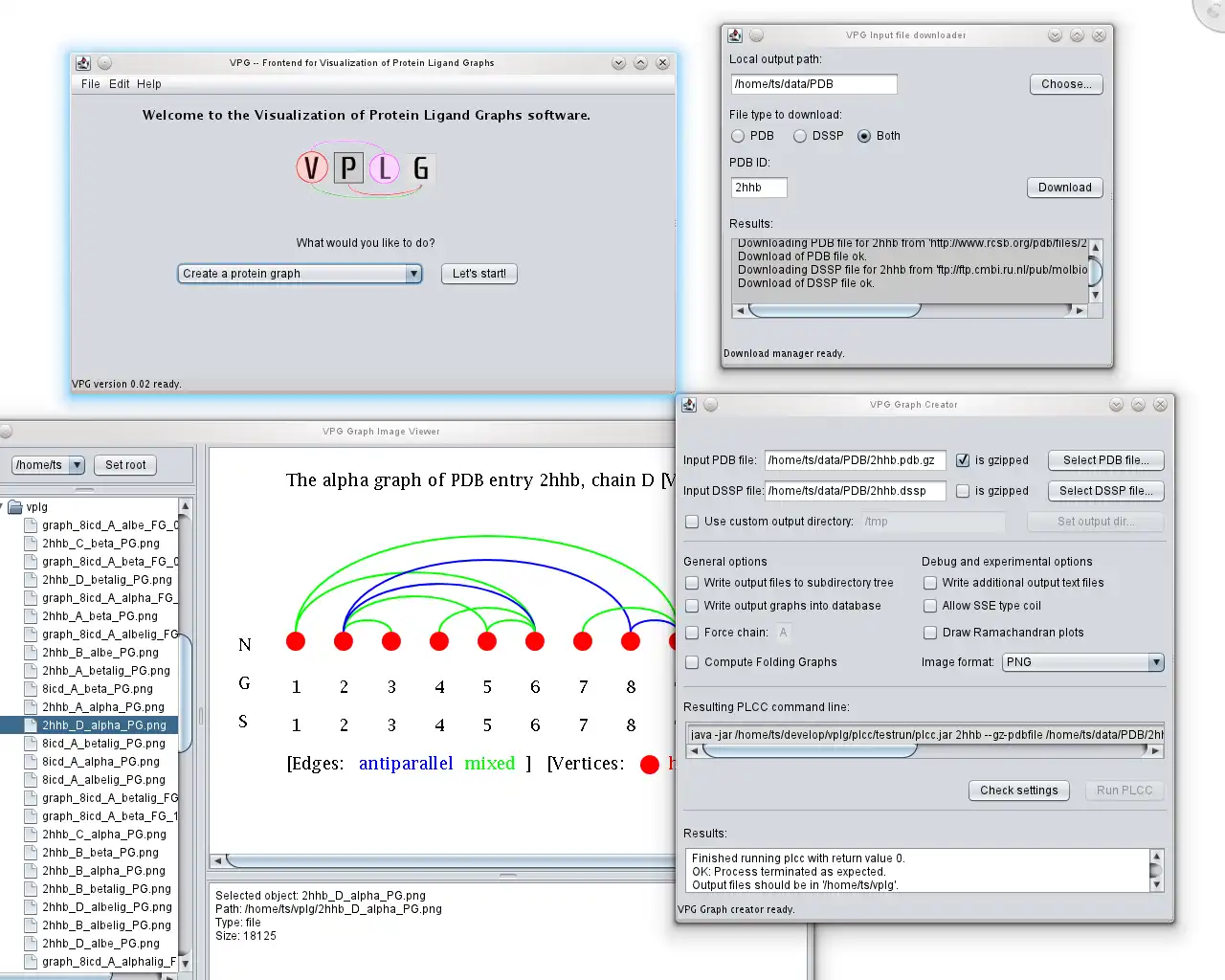
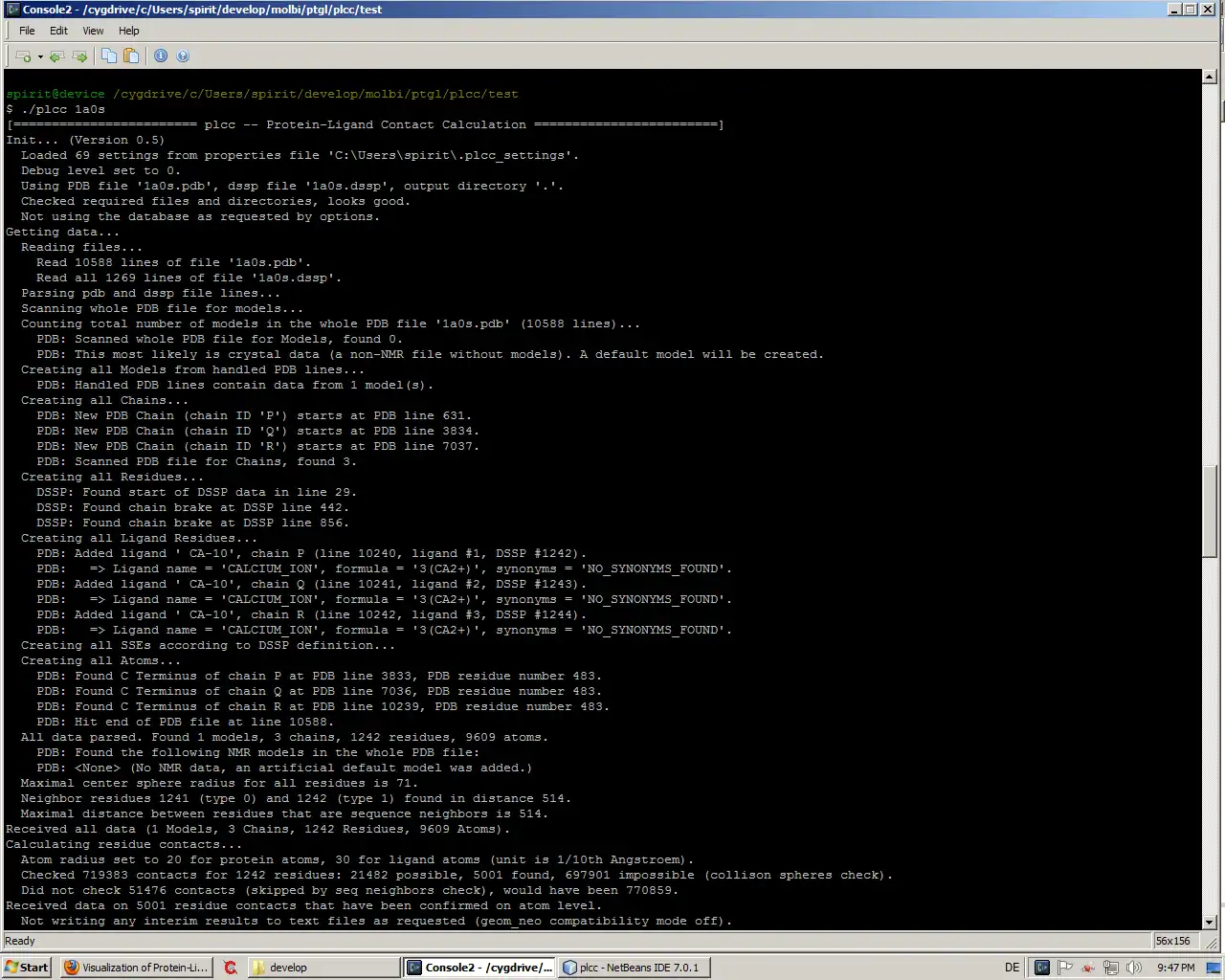
SCREENSHOTS
Ad
Visualisierung von Protein-Ligand-Diagrammen zur Ausführung unter Windows online über Linux online
BESCHREIBUNG
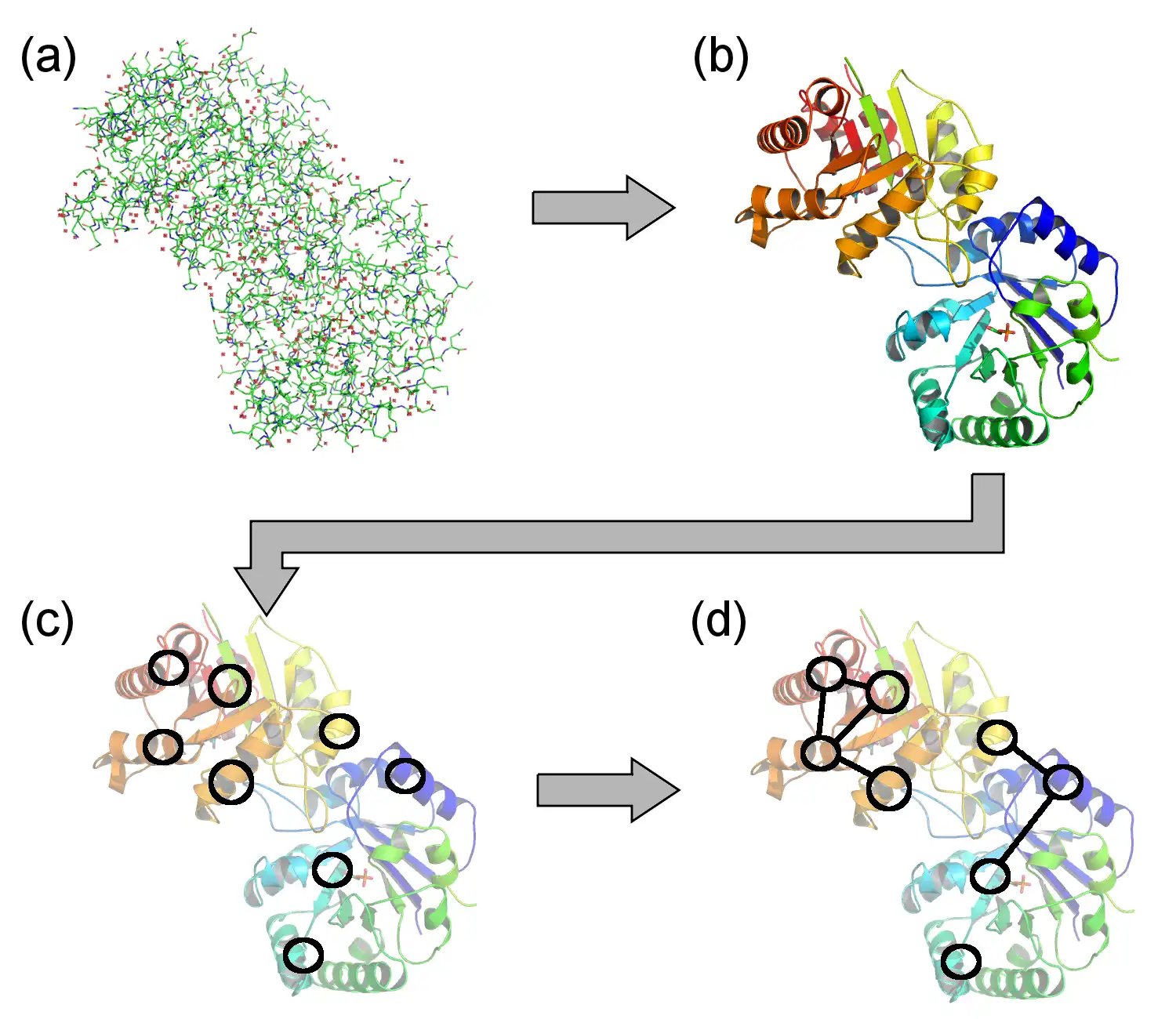
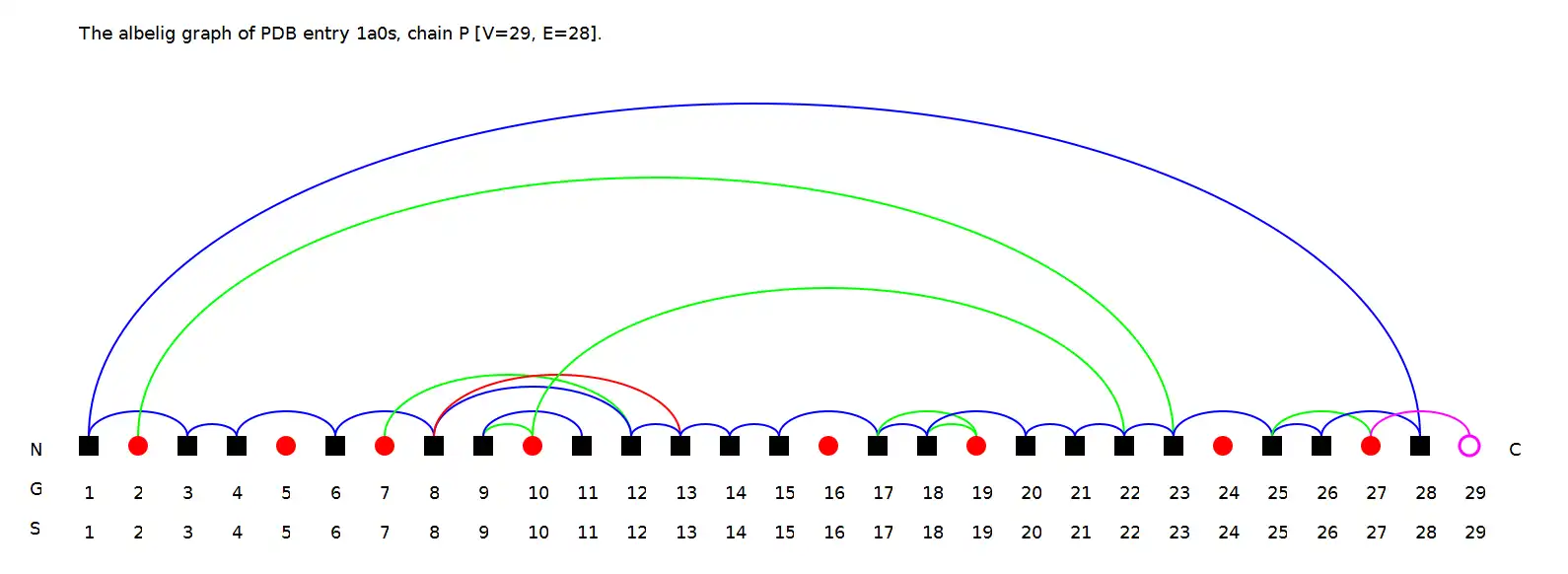
Das Softwarepaket Visualization of Protein-Ligand Graphs (VPLG) berechnet und visualisiert Proteindiagramme. Es arbeitet auf der Supersekundärstrukturebene und nutzt die Atomkoordinaten aus PDB-Dateien und die SSE-Zuweisungen des DSSP-Algorithmus.VPLG ist eine Befehlszeilensoftware. Wenn Sie nicht gerne Befehle eingeben, probieren Sie unseren PTGL-Webserver aus: http://ptgl.uni-frankfurt.de/
Eigenschaften
- Liest dreidimensionale Atomdaten aus PDB-Dateien und Sekundärstrukturzuweisungen aus DSSP-Dateien
- Berechnet aus den Daten ein Protein-Ligand-Diagramm und visualisiert das Diagramm
- Unterstützt die Ausgabe der Diagrammbilder in den Formaten Bitmap (.png) und Vektor (.svg).
- Exportiert Protein-Ligand-Diagramme in eine reine Textdatei in einem eigenen Format (.plg), das mit einem Computerprogramm einfach bearbeitet, analysiert und generiert werden kann, sowie in eine Reihe von Standard-Grafikdateiformaten
- Kann Proteindiagramme aus .plg-Dateien lesen und direkt visualisieren
- Kostenlose Open-Source-Software (FOSS)
- Datenbankunterstützung (völlig optional, standardmäßig kein Datenbankserver erforderlich)
- Die Befehlszeilenschnittstelle ermöglicht eine einfache Stapelverarbeitung über Shell-Skripte
- Ermöglicht das Filtern von Liganden nach Atomzahl
- Neue in Java geschriebene GUI, keine Notwendigkeit mehr, Konsolenbefehle einzugeben!
- Die Software, die den PTGL-Webserver betreibt: http://ptgl.uni-frankfurt.de/
Publikum
Wissenschaft/Forschung, Bildung, fortgeschrittene Endbenutzer
Benutzeroberfläche
Java Swing, Konsole/Terminal, Befehlszeile
Programmiersprache
Javac
Datenbankumgebung
JDBC
Dies ist eine Anwendung, die auch von https://sourceforge.net/projects/vplg/ abgerufen werden kann. Es wurde in OnWorks gehostet, um es auf einfachste Weise online über eines unserer kostenlosen Betriebssysteme ausführen zu können.